Identifican moléculas implicadas en la fisiología y patogenia de P. salmonis
Chile: Se trata de los llamados “ARNs no codificantes” y que fueron estudiados por científicos de Chile y Canadá a partir de once cepas del patógeno.
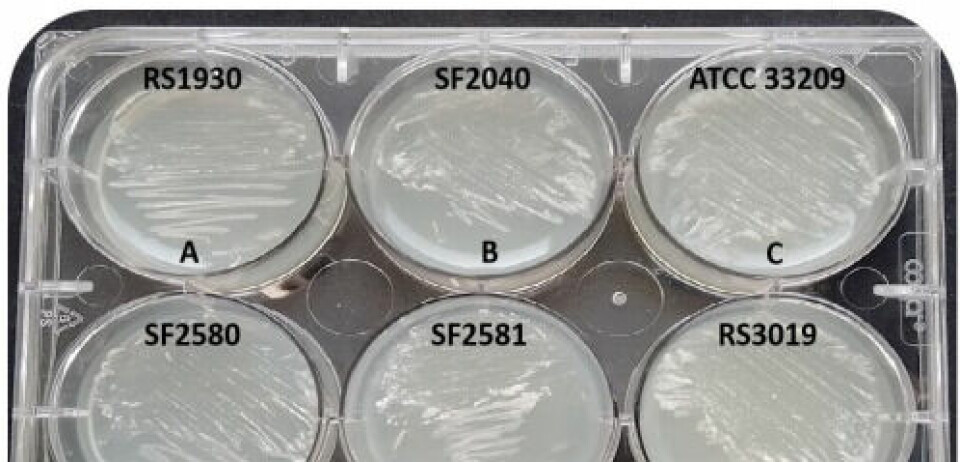
El conocimiento sobre los mecanismos reguladores de la patogénesis y fisiología de P. salmonis todavía es limitado debido a su difícil cultivo in vitro y diferencias genéticas entre genogrupos.
Los ARNs no codificantes (ncRNAs) son moléculas funcionales de ARN que no se traducen en proteína y que hoy en día se sabe que son importantes reguladores postranscripcionales de procesos fisiológicos bacterianos, así como también, de la respuesta a la temperatura, comunicación bacteriana, formación de biopelículas, metabolismo del hierro, virulencia y patogenia.
Para el caso de P. salmonis, el repertorio y las funciones que cumplen los ncRNAs no ha sido descrito.
Así, investigadores chilenos y canadienses publicaron recientemente un paper en donde definieron todos los ncRNAs comunes para once genomas secuenciados del patógeno y su posible papel en la regulación genética, patogénesis y fisiología bacteriana.
Encontraron que 1813 ncRNAs conformaban el “repertorio” de P. salmonis y que, de estos, 1383 estaban presentes en todos los genomas representando el core o núcleo de ncRNAs.
“Esta es la primera descripción de los ncRNA presente en el genoma de P. salmonis. Las diferentes familias de ncRNA presentes en diferentes aislados de la bacteria podrían utilizarse para determinar el origen geográfico, la virulencia de un aislado específico o como blancos para nuevos tratamientos antibacterianos” expusieron los autores.
Por ejemplo, el ncRNA “Sx4” identificado en P. salmonis podría regular la expresión de la enzima arginina descarboxilasa, que desempeña un papel esencial en la colonización tisular y la resistencia a los ácidos durante la patogénesis en Escherichia coli enterohemorrágica y Shigella flexneri.
Sin embargo, los investigadores señalan que todas las funciones previstas de los ncRNA identificados y los circuitos regulatorios en la bacteria necesitan validación experimental.
“Desafortunadamente, las herramientas genéticas para P. salmonis aún no están desarrolladas para generar mutantes y así probar los efectos sobre la fisiología y patogenicidad. Sin embargo, a pesar de esta falta de herramientas genéticas, existe una validación funcional de los genes predichos a través de la expresión heteróloga. Estos ensayos podrían ser un buen enfoque para probar nuestras predicciones, especialmente para analizar la función mediante la estructura secundaria conservada en los ncRNAs de P. salmonis” concluyeron.
Revise el abstract del estudio aquí