Modelo genómico esclarece funciones metabólicas de P. salmonis
Chile: Investigadores de Chile y Francia presentaron un nuevo modelo metabólico a escala genómica construido a partir de la cepa LF-89 de Piscirickettsia salmonis que ayuda a una mejor comprensión de las rutas metabólicas y diferenciación filogenética de la bacteria.
Hace más de dos décadas, se aisló la cepa LF-89 de P. salmonis, la primera cepa conocida de este patógeno. Desde entonces, se han desarrollado numerosos estudios para dilucidar aspectos biológicos y patogénicos que siguen sin ser resueltos, dificultando el establecimiento de estrategias efectivas contra el SRS.
Las distintas cepas chilenas se organizan filogenéticamente en dos grupos, G1 (LF-89) y G2 (EM-90), presentando características fenotípicas distintas.
Una forma sistemática de estudiar el metabolismo de un organismo es mediante el desarrollo de un modelo metabólico a escala genómica (GSM) y el uso del análisis basado en restricciones. Los GSM se generan a partir de la reconstrucción detallada de una red metabólica del organismo.
Estas reconstrucciones se basan en proteínas y genes metabólicos que son bases de datos de las funciones metabólicas. Además, los GSM son una representación matemática de estas redes reconstruidas que incluyen la adición de restricciones fisicoquímicas y ambientales. Estos modelos se pueden usar para predecir fenotipos metabólicos in silico bajo diferentes escenarios de crecimiento, así como las características esenciales de los genes y los metabolitos.
Modelo metabólico
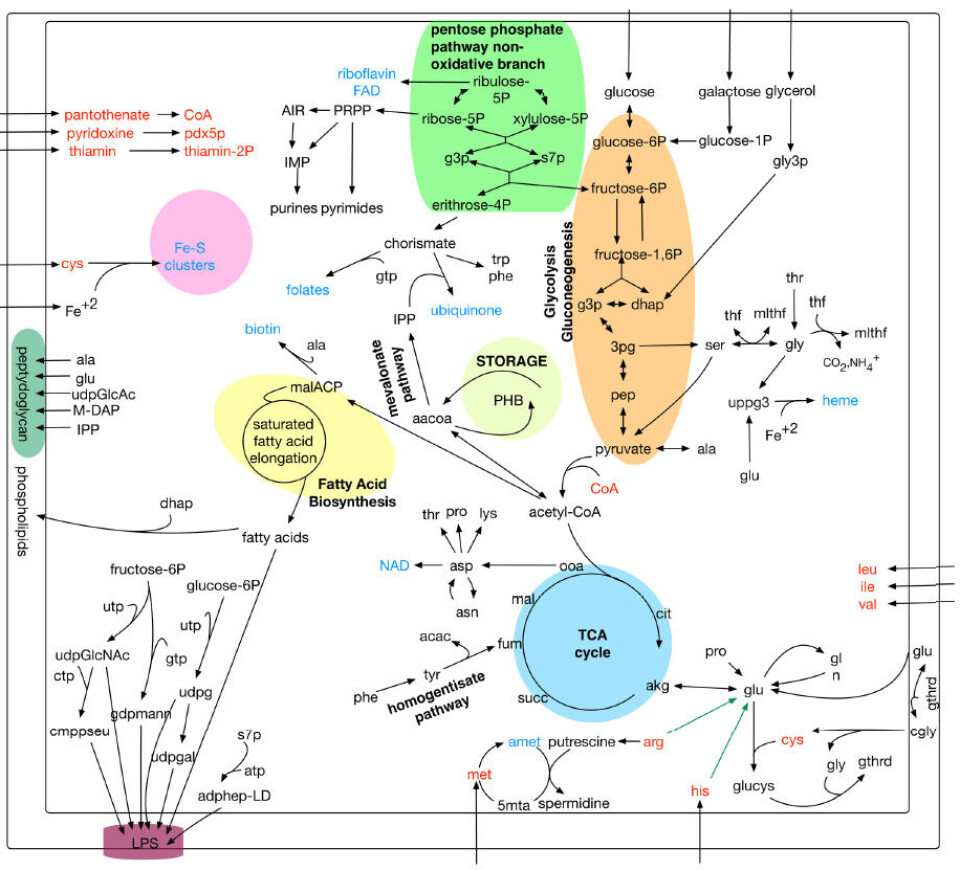
Recientemente se presentó un primer modelo metabólico (iPF215) para la cepa LF-89, que representa 215 de los genes del patógeno (Fuentealba et al., 2017). Sin embargo, diversos investigadores de la Universidad de Chile, Universidad Adolfo Ibañez, Centro FONDAP de Regulación del Genoma (CRG) y la Universidad de Rennes (Francia), publicaron un trabajo en la revista Frontiers in microbiology donde presentaron un nuevo GSM más completo de la misma cepa.
El nuevo modelo tiene más del doble de genes y reacciones comparado con el anterior, incluyendo 584 genes que representa el 19% de los genes en el genoma de la cepa LF-89, 801 metabolitos únicos y un total de 1.323 reacciones. Entre estas reacciones, el 80% tiene una asociación genética. Las reacciones sin un gen vinculado a ellas, son principalmente reacciones de intercambio de transporte o sintéticas
Dentro de los resultados de los diversos ensayos, los autores descubrieron que P. salmonis presenta una menor flexibilidad en la red metabólica que otros patógenos. También determinaron los metabolitos esenciales necesarios para el crecimiento de la bacteria y demostraron que puede beneficiarse de diferentes fuentes de carbono.
Por último, mediante la construcción de un modelo para la cepa A1-15972 (EM-90) y posterior comparación de ambos modelos con el pangenoma de P. salmonis, los autores demostraron que los genes metabólicos descritos en los modelos no solo confirman la separación de ambas cepas, sino que también, la existencia de los dos grupos filogenéticos.
Como conclusión los expertos dijeron que “ambos modelos constituyen una base de conocimiento para el metabolismo de P. salmonis y pueden ser usados para guiar el cultivo eficiente de la bacteria y también, para la identificación de objetivos farmacológicos específicos” los que podrían ayudar al control del patógeno.
Lea el artículo completo aquí

















