Perspectivas del conocimiento en la era genómica de Piscirickettsia salmonis
Jörn Bethke1,2, Jaime Figueroa2,3, Alejandro Yáñez2,3 & Ruben Avendaño-Herrera1,2 1 Laboratorio de Patología de Organismos Acuáticos y Biotecnología Acuícola, Facultad de Ciencias Biológicas, Universidad Andrés Bello. 2 Interdisciplinary Center for Aquaculture Research, Fondap Incar. 3 Instituto de Bioquímica y microbiología, Facultad de Ciencias, Universidad Austral de Chile.
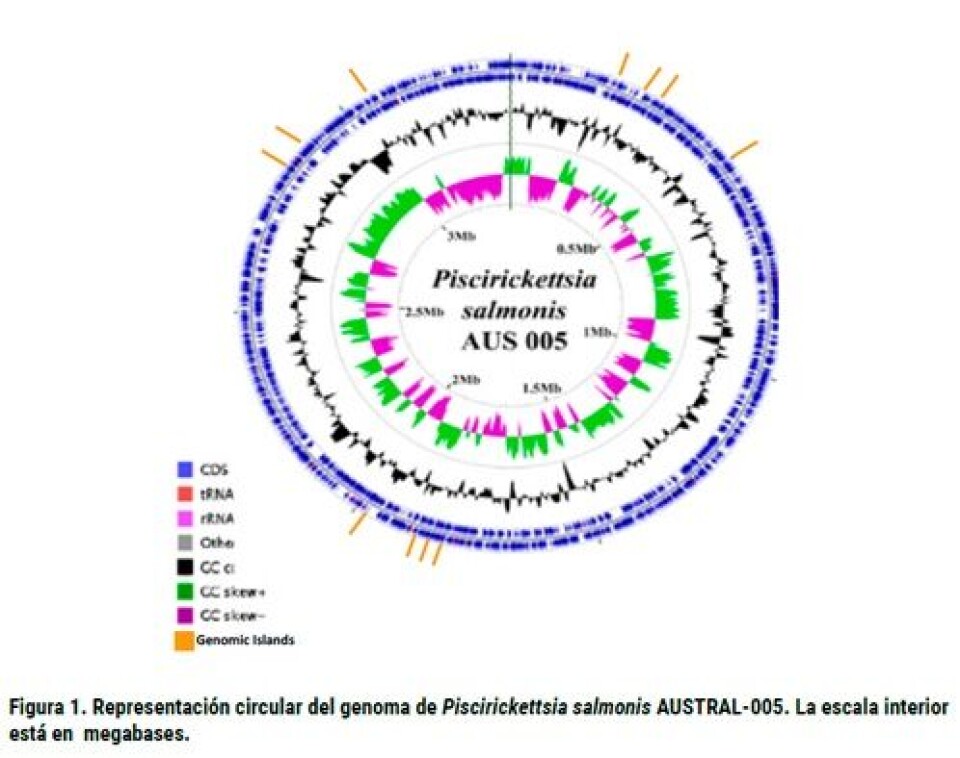
La Piscirickettsiosis es una enfermedad infecto-contagiosa que puede tener un curso agudo o crónico y sus hospederos principales corresponden a especies salmónidas cultivadas en Chile. El primer brote de esta enfermedad se remonta a 1989, cuando causó masivas mortalidades de ejemplares de salmón Coho (Oncorhynchus kisutch) mantenidos en engorda en el estero de Huito. El desconocimiento del agente etiológico causante de signos clínicos macroscópicos como esplenomegalia en diferentes grados y nódulos blanquecinos en el hígado de los peces (Fryer y col., 1990), provocó que la infección fuera llamada “Síndrome de Huito”. Poco tiempo después se reportaron mortalidades en las otras dos especies de salmónidos cultivados bajo las mismas condiciones (Cvitanich y col., 1991). En estos casos, el patógeno aislado desde peces infectados se identificó como una bacteria intracelular similar a una rickettsia y, por ello, la patología fue erróneamente denominada “Síndrome Rickettsial del Salmón (SRS)”. Sin embargo, el análisis de la secuencia del ADNr del gen 16S de la bacteria permitió identificar como agente causal una bacteria fastidiosa Gram-negativa aeróbica perteneciente al orden Thiotrichales denominada Piscirickettsia salmonis nov., sp. nov. (Fryer y col., 1992). Además, el análisis de este gen como herramienta taxonómica permitió incluir a P. salmonis en la clase de Gamma proteobacterias, la cual está estrechamente relacionada con Francisella sp., Legionella y Coxiella, distinto a los miembros de Rickettsia sp., las cuales pertenecen a la clase de las alfa proteobacterias (Fryer y col. 1992). Desde entonces, la distribución del patógeno se ha ampliado, siendo descrito en otras áreas geográficas distintas de Chile. Sin embargo, en la actualidad causa masivas mortalidades sólo en la industria chilena, especialmente en salmón Atlántico (Salmo salar). Así, esta especie registra un porcentaje de mortalidad cercano al 19% en promedio en la industria. De este porcentaje, el 25% corresponde a enfermedades, de las cuales casi 80% está asociado con Piscirickettsiosis. Sin duda, el Programa Sanitario Específico de Vigilancia y Control de Piscirickettsiosis ha sido una herramienta útil en la detección temprana y en el seguimiento que realiza el Servicio Nacional de Pesca y Acuicultura a los casos en campo; y así controlar los brotes y evitar la diseminación del patógeno. Además, la recopilación de datos epidemiológicos y las fiscalizaciones permiten focalizar los esfuerzos de prevención y control en las poblaciones de mayor riesgo. Hasta ahora, las causas de la mayor virulencia de los aislados de P. salmonis en los peces cultivados en sistemas acuáticos marinos son desconocidas, aunque existe un acuerdo tácito que es multifactorial. El objetivo de este artículo es profundizar en el conocimiento de la diversidad existente dentro de la especie P. salmonis, focalizándonos especialmente en los componentes moleculares y genómicos. Existen numerosas herramientas moleculares que permiten estudiar la variabilidad intra-específica en bacterias, las cuales van desde comparación de los patrones de amplificación generados por métodos de PCR (e.g. RAPD, ERIC, REP, etc.), restricción del genoma del microorganismos y, más recientemente, la secuenciación de genes constitutivos y que determinan funciones metabólicas en la bacteria, como la tipificación de secuencias de multilocus (MLST). Cada una de estas técnicas tienen sus ventajas y desventajas. Los primeros estudios en P. salmonis se remontan a 1999, cuando Mauel y colaboradores analizaron la secuencia transcrita interna del espaciador entre genes rRNA 16S y 23S (ITS) y describen la existencia de dos grupos filogenéticos, reconociendo como cepas representativas de cada grupo la cepa de salmón Coho LF-89 (actualmente cepa tipo de la especie) y la cepa de salmón Atlántico EM-90. Por lo tanto, Casanova y col. (2001) señalan que P. salmonis tiene una región ITS A y otra B. En cuanto a la virulencia de cada uno de estos grupos, sin duda un aporte inicial es el realizado por House y colaboradores en 1999, quienes reportan diferencias en la virulencia en los aislados obtenidos en distintos países y también diferencias en su ITS como Chile (LF-89), Canadá (ATL-4-91) y Noruega (NOR-92). Rojas y col. (2008) caracterizaron la secuencias de ITS de una variante pequeña e infectiva de P. salmonis (sP.s) recuperada de células CHSE-214 infectadas y desde peces infectados de forma natural. La región ITS de la variante fue diferente a aquella de la cepa LF-89 a partir del cual se originó la variante. Estos autores demuestran que sP.s es totalmente activa, con una estructura celular más pequeña (<0,2 μm) que el rango de tamaño de la bacteria estándar (0,5–1,5 μm) y la progenie en condiciones in vitro de la variante parece ser idéntica a la progenie de la cepa LF-89. Aún cuando algunos grupos de investigación en los últimos años han propuesto la existencia de una nueva especie bacteriana, sP.s es una variante infecciosa de la cepa tipo y no una nueva cepa, la que se originó probablemente como resultado de una estrategia de supervivencia de la bacteria en respuesta a la limitación de las condiciones de crecimiento. Incluso, esta variante puede ser responsable de la infección horizontal en agua de mar (Rojas y col., 2008). Más recientemente, estudios realizados por el Dr. Jaime Figueroa empleando el mismo gen, pero un universo de más de 50 aislados de P. salmonis, representativos de los últimos cinco años ha señalado la existencia de cuatro genogrupos o Clúster. A partir de cada genogrupo, se logró secuenciar parcialmente los genomas de 10 cepas del patógeno, pertenecientes a los cuatro genogrupos en que se distribuyen las cepas nacionales. Los genogrupos muestran cepas similares a LF-89, otro con cepas similares a EM-90, y los dos restantes con cepas con características génicas diferentes de las dos cepas más ancestrales. Los genomas muestran características claras que permiten clasificar a P. salmonis como una bacteria intracelular facultativa. Adicionalmente, se logró definir cerca de 3.500 regiones codificantes correspondientes a genes de diversas categorías de funciones. En tanto, experimentos de electroforesis de campo pulsado (PFGE), muestran que el tamaño aproximado del genoma de esta bacteria es cercano a 4,3 millones de pares de bases (pb), sin la presencia aparente de plásmidos, al menos por esta metodología. Estos estudios genómicos se han complementado con análisis proteómicos comparativos entre cepas de varios genogrupos. Todos muestran una gran variabilidad en los patrones de proteínas mostrados, dando cuenta de que probablemente la variabilidad a este nivel, es aún más grande que la mostrada inicialmente por la genómica del cistrón ribosomal. Aunque existen otros estudios usando distintas herramientas moleculares, los resultados de diversidad intra-específica no han sido publicados en revistas científicas de corriente principal, por lo cual, han sido omitidos de este artículo. La secuenciación del genoma completo de bacterias ha entregado nuevas métricas taxonómicas, incluyendo el promedio de identidad de nucleótidos (ANI, sigla en inglés), calculado a partir de la comparación de todas las secuencias compartidas entre las dos cepas. Esta herramienta puede ser empleada incluso para la descripción de nuevas especies bacterianas, ya que el ANI presenta una fuerte correlación con los valores de hibridación ADN-ADN (DDH), pues con un valor de ANI ≥ 95% es equivalente al tradicional 70% de umbral de DDH (Goris y col., 2007). Para P. salmonis, actualmente se encuentran disponible siete genomas en la base de datos GenBank (www.ncbi.nlm.nih.gov). Sin embargo, sólo cuatro de ellos han sido publicados en Genome Announcements y corresponden al genoma de la cepa tipo LF89 (Eppinger y col., 2013), cepa Austral-005 (Yáñez y col., 2014a) y los aislados A1-15972 y B1-32597 (Bohle y col., 2014), seleccionados como representantes del tipo genómico EM-90 y LF-89, respectivamente. En las tablas 1 y 2 se muestran las estadísticas globales en relación a la tecnología empleada en la secuenciación, así como el ensamble y el informe de anotación de los distintos genomas de P. salmonis. Es interesante señalar que de los siete genomas secuenciados, tres de ellos corresponden a la secuenciación realizada del ADN obtenida de la cepa tipo LF-89 (equivalente a la cepa ATCC VR-1361), los cuales mostraron un tamaño total aparente entre 2.805.478 y 3.068.090 pb, mientras que la cepa EM-90 tiene un tamaño aparente de 3.035.439 pb. Es interesante señalar que P. salmonis Austral-005 aislada de trucha arcoíris (Oncorhynchus mykiss), A1-15972 de salmón Atlántico y B1-32597 de salmón Coho tienen un tamaño aparente mayor de genoma, variando entre 3.138.697 a 3.529.9595 bp. Por lo tanto, si consideramos los resultados de PFGE, aún quedan por secuenciar aproximadamente 1 millón de pb para cerrar el genoma más completo de P. salmonis correspondiente al aislado Austral-005 (Figura 1). Por el contrario, el contenido de guanina y citosina (%GC), una característica propia del genoma de un organismo bacteriano como P. salmonis, el valor es muy similar con 39,88 ± 0,22. El análisis del contenido de genes y relaciones genéticas existentes en P. salmonis pueden ayudar a mejorar nuestra comprensión de la biología, evolución y adaptación al ambiente del patógeno, así como conocer las rutas metabólicas y el repertorio de factores de virulencia presentes en la bacteria. Además, la información puede ser empleada para el desarrollo de herramientas biotecnológicas que permitan de forma directa o indirecta la prevención y control de la Piscirickettsiosis a corto plazo. Por ejemplo, el análisis de las rutas metabólicas del genoma del aislados Austral-005 permitió el desarrollo de un medio de cultivo líquido libre de células, denominado Austral-SRS Broth, el cual está suplementado con cisteína y sales de fierro, componentes imprescindibles que permiten el crecimiento de P. salmonis, considerada tradicionalmente como un patógeno intracelular estricto (Yáñez y col., 2012). Además, el medio ha permitido el estudio de susceptibilidad a drogas en condiciones in vitro (Yáñez y col., 2014b) así como ser empleado para el desarrollo de vacunas. Respecto a mecanismos de virulencia, existe concordancia en los tres artículos publicados que P. salmonis presenta el sistema de secreción de tipo IV (T4SSs), el cual es un complejo multiproteico que juega un rol en la virulencia (Waksman & Orlova, 2014), incluyendo la supervivencia intracelular y/o la replicación de la bacteria. Recientemente, se informó de la expresión de genes relacionados con el T4SS en la cepa LF-89 (Gómez y col., 2013). Es interesante señalar que sólo tres T4SSs fueron encontrados en el genoma del aislado B1-32597, mientras que el genoma A1-15972 contiene dos T4SSs, faltando en B1-32597 una proteína de ~30 kb que lleva un T4SS completo. A su vez, el análisis de Austral-005 (Yáñez y col., 2014a) mostró dos sistemas putativo toxina-antitoxina (TA), higAB y txe/yoeB, y cuatro genes putativos que codifican proteasas (clp, lon, M22 y M48). El análisis de la secuencia del genoma también reveló la presencia de los sistemas de secreción tipo I, II y IV así como seis genes putativos que codifican proteínas de choque térmico (HSP) (Hsp33, GroE, Hsp90, DnaJ, Hsp70, Hsp20 y). En cuanto a metabolismo del hierro y su transporte, se presentan cuatro genes putativos que codifican proteínas relacionadas con sideróforos y proteínas receptoras. Sorprendentemente, el genoma de esta bacteria inmóvil reveló en todos los casos la presencia de genes que codifican componentes de flagelos, por lo que desde un punto de vista biológico evolutivo es interesante conocer si dependiendo de las condiciones ambientales P. salmonis es capaz de presentar y emplear un orgánulo similar a un flagelo. Basado en los antecedentes descritos anteriormente, la era de la genómica llegó para quedarse, ya que la información obtenida de los genomas ha permitido avanzar mucho más rápido en el conocimiento integral de P. salmonis, así como corroborar con una mirada más molecular numerosos resultados funcionales publicados antes del año 2013. Por lo tanto, el adecuado uso de la información genómica proyecta un futuro esplendoroso en el conocimiento mas profundo de P. salmonis. Sin embargo, es imprescindible aunar los esfuerzos para cerrar al menos un genoma del patógeno, sea la cepa tipo LF-89 o el aislado con mayor tamaño de secuencia aparente que permita emplearlo como un genoma modelo. En tanto, obtener el genoma de otros aislados de campo que representen la estructura poblacional de P. salmonis, considerando hospedero, año de aislamiento, área geográfica y aspecto patológicos son clave, ya que podrían proporcionar antecedentes relevantes asociados con virulencia, patogenicidad y resistencia a antibióticos, lo cual no podrá ser respondido con un único genoma.
Agradecimientos Conicyt/Fondap/15110027 de la Comisión Nacional de Investigación Científica y Tecnológica (Conicyt, Chile).
Referencias Bohle y col., (2014) Comparative genome analysis of two isolates of the fish pathogen Piscirickettsia salmonis from different host revealed major differences in virulence-associated secretion system. Genome Announcements 6, e01219-14. Casanova y col., (2001) tRNA genes were found in Piscirickettsia salmonis 16S–23S rDNA spacer region (ITS). FEMS Microbiology Letters 197, 19–22. Cvitanich y col., (1991) The isolation of a rickettsia-like organism causing disease and mortality in Chilean salmonids and its confirmation by Koch’s postulate. Journal of Fish Diseases 14, 121–145. Eppinger y col., (2013) Draft genome sequence of the fish pathogen Piscirickettsia salmonis. Genome Announcements 6, e00926-13. Fryer y col., (1990) Isolation of a rickettsiales-like organism from diseased Coho salmon (Oncorhynchus kisutch) in Chile. Fish Pathology 25, 107–114. Fryer y col., (1992) Piscirickettsia salmonis gen. nov.; sp. nov. the causative agent of an epizootic disease in salmonid fishes. International Journal of Systematic Bacteriology 42, 120–126. Gómez y col., (2013) Evidence of the presence of a functional Dot/Icm type IV-B secretion system in the fish bacterial pathogen Piscirickettsia salmonis. PLoS One 8(1):e54934 Goris y col., (2007) DNA–DNA hybridization values and their relationship to whole- genome sequence similarities. International Journal of Systematic Evolutionary Microbiology 57, 81–91. House y col., (1999) Relative virulence of three isolates of Piscirickettsia salmonis for Coho salmon Oncorhynchus kisutch. Diseases of Aquatic Organisms 35, 107–113. Mauel y col., (1999) Phylogenetic analysis of Piscirickettsia salmonis by 16S internal transcribed spacer (ITS) and 23S ribosomal DNA sequencing. Diseases of Aquatic Organisms 35, 115–123. Rojas y col., (2008) Characterization of a novel and genetically different small infective form of Piscirickettsia salmonis. Microbial Pathogenesis 44, 370–378. Yáñez y col., (2012) Broth medium for the successful culture of the fish pathogen Piscirickettsia salmonis. Diseases of Aquatic Organisms 97, 197–205. Yáñez y col., (2014a) Draft genome sequence of virulent strain AUSTRAL-005 of Piscirickettsia salmonis, the etiological agent of Piscirickettsiosis. Genome Announcements 2, e00990-14. Yáñez y col., (2014b) Broth microdilution protocol for minimum inhibitory concentration (MIC) determinations of the intracellular salmonid pathogen Piscirickettsia salmonis to florfenicol and oxytetracycline. Journal of Fish Diseases 37, 505–509.