Proyecto descifra aspectos claves de Piscirickettsia salmonis
Chile: Desde el descubrimiento de nuevos genogrupos del patógeno hasta posibles reservorios, genoma, resistoma y marcadores genéticos de patogenicidad y virulencia, fueron algunos de los resultados obtenidos por la iniciativa.
"Olvidémosnos que es una rickettsia, no se parece en nada a una rickettsia, es más, es una gamaproteobacteria mucho más parecida a una Legionella o Francisella", comenzaba diciendo el Dr. Sergio Marshall en su presentación del pasado jueves en el worshop donde se dieron a conocer los resultados finales del proyecto FIE “Estudio integral de las bases biológicas y moleculares del ciclo de vida de Piscirickettsia Salmonis en el contexto de una aproximación epidemiológica para desarrollar estrategias que permitan su control” que encabeza.
Con una duración real de 13 meses y una colaboración de 15 investigadores chilenos y 5 alemanes, el proyecto dividido en cuatro paquetes de trabajo (WP) presentó interesantes resultados y descubrimientos en torno al patógeno intracelular.
Haciendo énfasis en el hecho de que con la evidencia presentada hay que cambiar la forma en cómo se miraba y estudiaba la bacteria, el académico e investigador de la Pontificia Universidad Católica de Valparaíso (PUCV), planteó que lo que verdaderamente ocurre en el pez es un consorcio bacteriano, donde hoy ya se sabe que existe más de una variante de los dos genogrupos reconocidos como la base de la Piscirickettsiosis en Chile.
Así, señaló que con la información obtenida de los distintos WP pudieron definir, entre otros, marcadores asociados a la patogenicidad y virulencia de la bacteria para cumplir con dos propósitos: encontrar mecanismos de detección clínica temprana y generar otras alternativas de prevención, detallando que “estamos produciendo estrategias para nuevas vacunas y vacunas vivas atenuadas, donde la edición génica surje como uno de los elementos primordiales”.
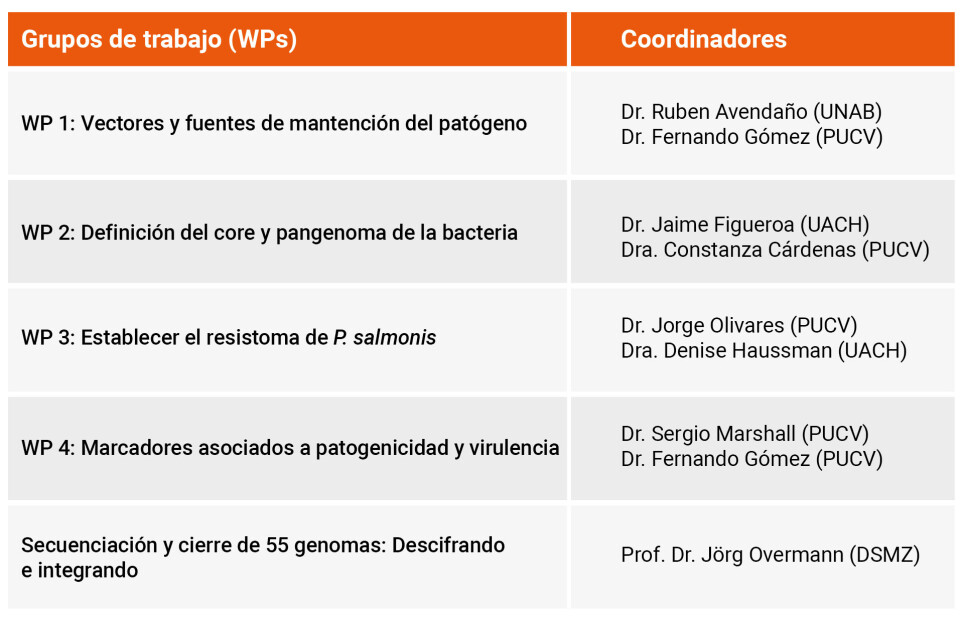
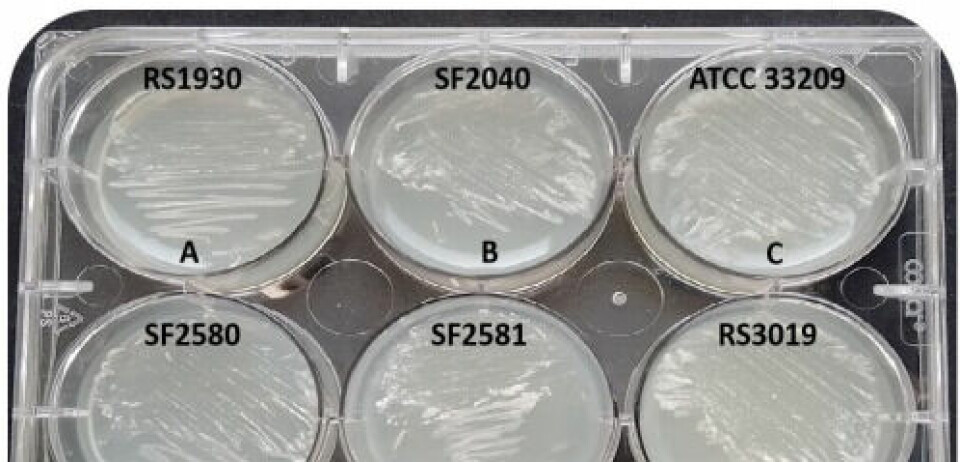
También, gracias a la integración de WP, lograron secuenciar y cerrar 55 genomas de P. salmonis, base de datos que estará disponible abiertamente para quien lo solicite y que representan información reunida durante 15-20 años.
Reservorio y antibióticos
Con la evidencia de que la bacteria logra sobrevivir y multiplicarse en protozoos de agua dulce y agua de mar, el experto recalcó la importancia de este hallazgo frente a la coinfección y mantención de la enfermedad en centros de cultivo.
“En agua de mar, se puede encontrar en más de un tipo de protozoo e interesantemente, se detectó P. salmonis en el protozoo Vanella sp., uno de los agentes causales de la enfermedad amebiana de las branquias (AGD). Esta asociación nos hace pensar que éste puede ser un mecanismo que utiliza la bacteria para ingresar al pez y dispersarse, donde además de ser una estrategia de supervivencia, podría explicar la transferencia entre ciclos de producción o diferentes centros de cultivo”, expuso.
Adicionalmente, en relación a la terapia con antimicrobianos, uno de los WP descubrió evidencia con respecto a que los antibióticos que se utilizan en la actualidad, aparentemente no tienen contacto con la bacteria dentro del pez. Lo anterior debido a que no encontraron pérdida de la susceptibilidad a un número significativo de peces analizados.
“Pensamos que el efecto del antibiótico sólo evita la infección horizontal de los peces, pero no mejora al pez enfermo, es un elemento que tenemos que considerar y discutir", aclaró el Dr. Marshall.
Asimismo, el equipo de trabajo logró identificar OMV´s bacterianos que podrían servir como marcadores de patogenicidad y miRNAs que serían útiles al momento de diseñar un sistema de detección temprana mínimamente invasivo, no letal y que complementaría la respuesta del pez a la eventual infección con el patógeno.
Próximos pasos
Por último, el académico de la PUCV indicó a Salmonexpert que el siguiente paso es dar a conocer al sector productivo que la inversión dio resultados significativos y que estas iniciativas deben seguir.
““El cómo lo hacemos presenta una dificultad, tenemos que tener un programa que se extienda. Algo concreto que quiero proponer es hacer un pioloto, para que en un brote de SRS podamos obtener una muestra directamente del pez, hacer la secuenciación y comparar los resultados con nuestra base de datos. Los resultados nos permitirán concretar investigaciónes que generen vacunas o técnicas diagnósticas, por ejemplo”, puntualiza Marshall, concluyendo: “recordemos la importancia de los resultados; ¿es un genogrupo? no, ya sabemos que son tres”.