Variación genética entre cepas de Flavobacterium psychrophilum aisladas de salmónidos de cultivo y nativos de Chile y Noruega
Patricia Apablaza1, Asbjørn Dyrkorn Løland1,*, Øyvind Jakobsen Brevik1,*, Pedro Ilardi2, Juan Battaglia2 y Are Nylund1 1 Fish Diseases Research Group, Departmento de Biologia, Universidad de Bergen, Bergen, Noruega 2 Veterquimica, Santiago, Chile Correspondencia: Patricia Apablaza, Fish Diseases Research Group, Department of Biology, University of Bergen, Norway, Post-Box 7803, 5020 Bergen, Norway. E-mail: Patricia.Apablaza@bio.uib.no * Estos autores contribuyeron igualmente a este estudio
Introducción Después de su primera descripción por Davis, 1946, en Estados Unidos, Flavobacterium psychrophilum se ha expandido en la acuicultura a nivel mundial (Borg, 1948), incluyendo Noruega (Duesund et al., 2010, Nilsen et al., 2011) y Chile (Bustos et al., 1995). La patología clínica causada por el patógeno se denominó clásicamente ‘enfermedad del pedúnculo’ que incluye lesiones ulcerativas en el pedúnculo del pez. Actualmente, los cuadros clínicos son conocidos como ‘rainbow trout fry syndrome’ (RFTS) y ‘bacterial cold water disease’ (BCWD). F. psychrophilum se ha aislado de diferentes hospedadores salmónidos y no-salmónidos, siendo descrita una cierta afinidad por la trucha arco iris. En los últimos años, F. psychrophilum ha emergido como un importante patógeno de peces, causando grandes pérdidas económicas a causa de las altas mortalidades y, además, por los costos asociados al incremento en el consumo de antibióticos para su tratamiento. Se ha descrito que este patógeno puede transmitirse tanto horizontal (Madetoja et al., 2000) como verticalmente (Cipriano & Holt, 2005). F. psychrophilum se ha aislado del interior y de la superficie de huevos de salmónidos, incluso después de la desinfección con iodóforos, desde fluidos ováricos y de gónadas de reproductores machos. Así, se ha sugerido que el movimiento de ovas provenientes de reproductores infectados con el patógeno, puede tener un rol importante en la transmisión transgeneracional y a largas distancias por medio del comercio internacional de ovas (Kumagai & Takahashi, 1997). La tipificación de cepas de patógenos bacterianos tiene un rol importante en la prevención de transmisión, tratamiento y para el diseño de estrategias de manejo (van Belkum et al., 2007), especialmente en la acuicultura. El genoma completo de F. psychrophilum fue publicado en el año 2007, permitiendo la comparación de genes asociados con virulencia, afinidad por diferentes hospedadores o distribución geográfica, entre diferentes cepas para el desarrollo de sistemas de genotipificación. Actualmente no hay descrito un sistema de tipificación adecuado y rápido para F. psychrophilum (Hesami et al., 2008). En 1998, Maiden y col. estableció el sistema de ‘multilocus sequence typing’ (MLST) como una herramienta adecuada para la tipificación de Neisseria meningitides, permitiendo analizar comparativamente resultados de diferentes laboratorios basados en las secuencias nucleotídicas de los patógenos. Desde entonces, MLST ha sido usado con éxito para diferenciar clones individuales intra-especies, determinando la variación de las secuencias nucleotídicas de 5-10 genes ‘housekeeping’ (HGs) (van Belkum et al., 2007). Comparando los tipos únicos de secuencias (ST) para cada cepa es posible generar ‘clusters’ en esquemas filogenéticos separando, por ejemplo, complejos clones virulentos de los no-virulentos. Nicolas y col. (2008) genotipificó 50 cepas de F. psychrophilum mediante MLST, que representan una distribución global incluyendo diferentes especies hospedadoras salmónidas y no-salmónidas, encontrando varios complejos clonales asociados a especies hospedadoras. Estos complejos clonales estaban distribuidos geográficamente en diferentes continentes, sugiriendo que esta conexión podría reflejar un nicho adaptativo especializado y/o rutas de transmisión preferentes. El objetivo del presente estudio fue describir mediante MLST y filogenéticamente las variaciones genéticas en una selección de siete HG en cepas de F. psychrophilum aisladas de salmónidos de cultivo en Chile y Noruega. Se incluyeron, además, cepas aisladas de salmónidos nativos de Noruega y secuencias genéticas de HG de las cepas descritas y depositadas en el GenBank por Nicolas y col. (2008). Con esto se espera comprender la posible variación genética entre las cepas de F. psychrophilum y la presencia de cepas idénticas, como una forma de asociar la acuicultura de dos regiones tan separadas geográficamente como lo son Chile y Noruega.
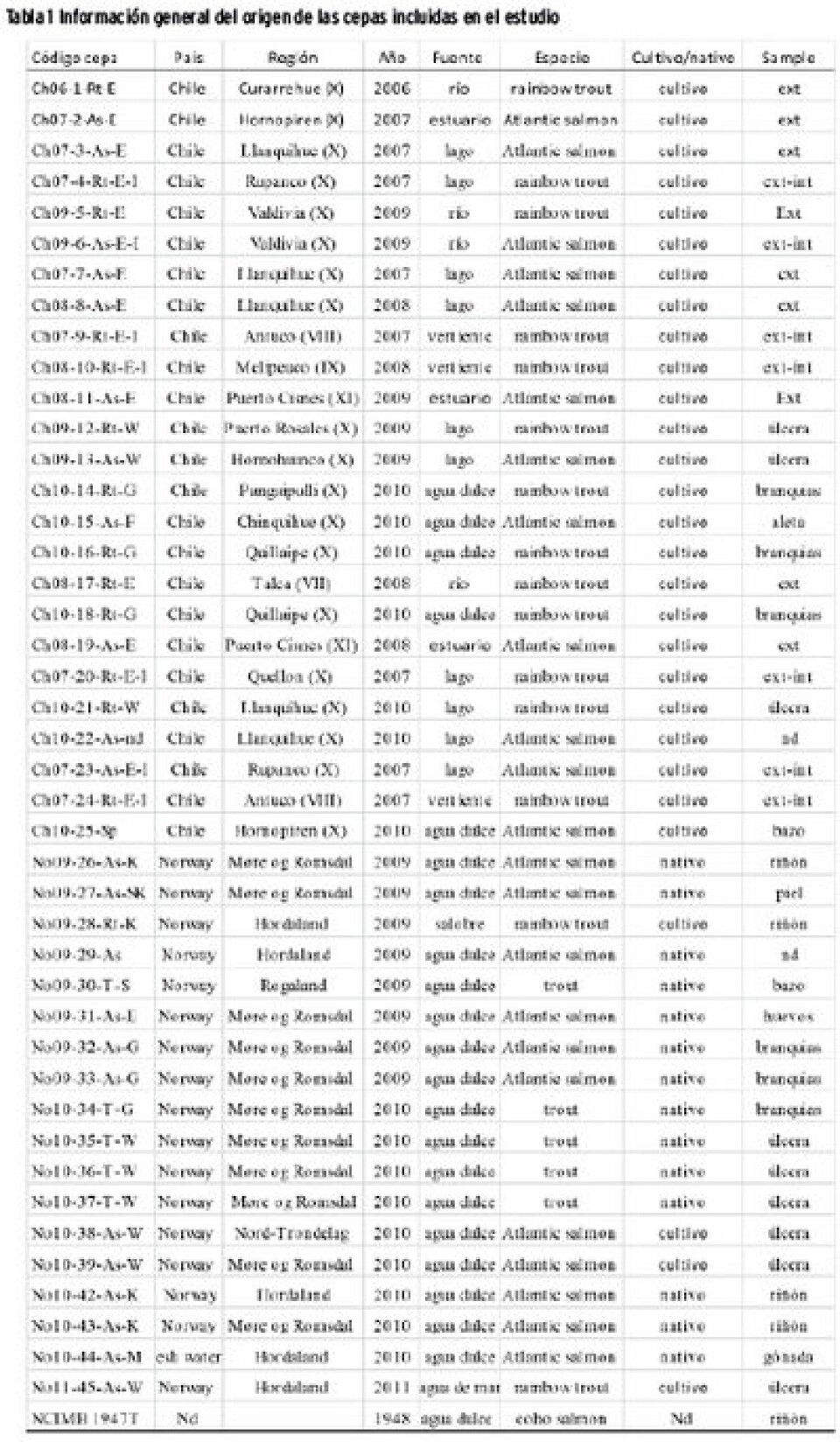
Materiales y métodos Aislamiemto de Flavobacterium psychrophilum Se incluyeron 44 cepas de F. psychrophilum aisladas desde el año 2006 hasta el 2011, 18 provenientes desde Noruega, 25 de Chile y la cepa tipo NCIMB 1947. Éstas se obtuvieron desde diferentes tejidos, como riñón, bazo, gónadas y desde los márgenes de úlceras en la piel (Tabla 1). La muestras se cultivaron en agar FLP y fueron incubadas a 15 °C por hasta tres semanas. Después de la caracterización fenotípica de las colonias en cultivo puro, la identificación de F. psychrophilum se realizó mediante la amplificación del gen 16S rRNA. Para la genotipificación de las cepas, fue extraído el DNA bacteriano desde cultivo puro usando el kit comercial DNeasy. Para las amplificaciones por PCR de los HG trpB, atpA, gyrB, fumC, murG, dnaK y tuf, se utilizaron partidores específicos para F. psychrophilum, basados en el estudio de Nicolas y col. (2008). Los productos de PCR fueron visualizados mediante electroforesis en gel de agarosa al 1%, purificados y secuenciados según lo descrito por Brevik y col. (2011). Las secuencias del gen 16S rRNA de F. columnare y F. frigidarium se incluyeron como ‘out-group’ en los análisis filogenéticos. Los alineamientos de los siete HGs fueron suplementados con secuencias homólogas obtenidas del GenBank para las 50 cepas de F. psychrophilum del estudio de Nicolas y col. (2008). El modelo evolutivo y las tasas de substitucion fueron calculados para todos los alineamientos usando el jModelTest. Los modelos filogenéticos para 16S rRNA y para los siete HGs fueron realizados usando el método Bayesian. Para el sistema MLST, los diferentes tipos de alelos (AT) dentro de los siete HGs fueron codificados como caracteres discretos (ej. 1–9, A–H). La base de datos (http://www.pasteur.fr/recherche/genopole/PF8/mlst/Flavopsy.html) para F. psychrophilum fue usada para codificar las secuencias homólogas en el set de datos del presente estudio. Los AT se usaron para crear las secuencias tipo (ST) de cada cepa. Con las STs se construyó un matriz de datos con el total de las 94 (44 del presente estudio más las 50 de Nicolas y col. (2008) de F. psychrophilum incluidas en este estudio.
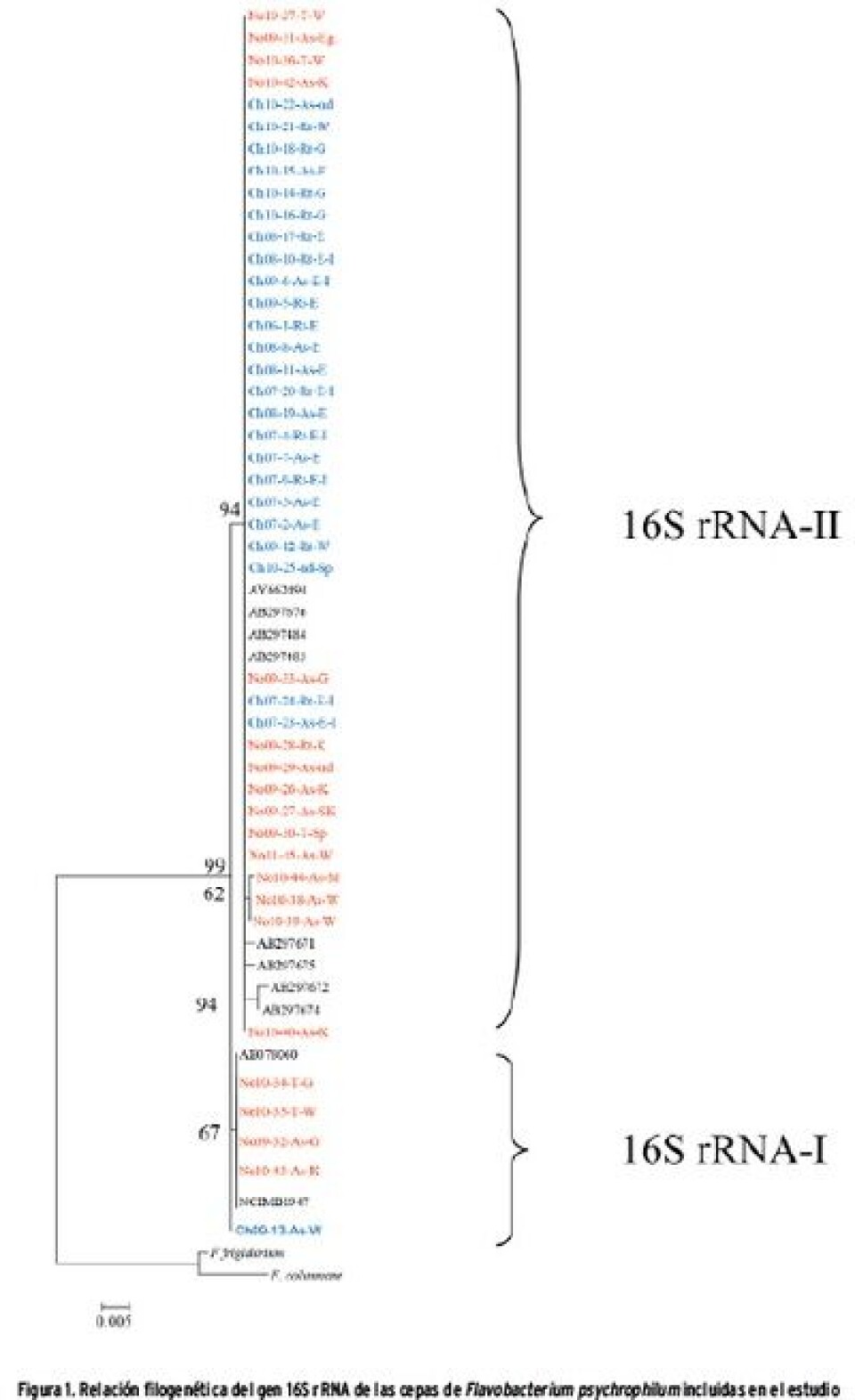
Resultados En los análisis filogenéticos del gen 16S rRNA, la resolución entre las secuencias fue relativamente baja, obteniéndose sólo dos clades (16S rRNA-I y 16S rRNA-II) (Fig. 1). La clade 16S rRNA-I, contiene siete secuencias con la especie tipo, incluidas dos obtenidas de salmón Coho en Estados Unidos. De las otras cinco cepas, cuatro son de salmón del Atlántico y trucha silvestre (Salmo trutta) en Noruega, mientras que la quinta es salmón del Atlántico en Chile. La clade 16S rRNA-II comprende las otras cepas conjuntamente con cepas de Japón. Los análisis filogenéticos de los HG tuf, murG, fumC, trpB, dnaK and atpA resultaron en sólo 2–3 clades, mientras que en gyrB se obtuvieron cinco clades (Fig. 2). Éstas no agrupan geográficamente las cepas, y en la mayoría de los casos contienen microorganismos de los diferentes continentes. Los grupos gyrB-I y gyrB-II incluyen sólo cepas noruegas obtenidas desde salmón del Atlántico de cultivo y nativos. Los resultados de MLST incluyen una combinación de los diferentes AT de los siete loci, que permiten distinguir 53 STs entre las 94 cepas (Fig. 3). De estas 53 STs, 38 fueron solamente muestreadas una vez. La ST6 obtuvo el más alto número de cepas que fueron sólo obtenidas en Europa (Francia, Suecia, Dinamarca y Noruega), desde 1988 hasta 2011. La ST9 contiene siete cepas de Chile, de las cuales cinco provienen de salmón del Atlántico, dos de trucha arcoíris y dos de salmón Coho. La ST20 contiene cinco cepas de salmón del Atlántico y trucha arcoíris de Chile y una de trucha arcoíris obtenida en Alemania. Dos STs, ST42 and ST49, contienen cinco cepas y cuatro aislados respectivamente, todas de Noruega. La ST42 contiene tres cepas de trucha y dos de salmón del Atlántico, mientras que las cepas de ST49 fueron todas obtenidas de salmón del Atlántico. El mayor número de cepas incluidas en este estudio proviene de Noruega (N = 19) y Chile (N = 28), las cuales se agrupan en 10 y 16 STs, respectivamente. Cepas de Noruega y Chile se encuentran en sólo una ST (ST36), en la cual ambos aislados son de trucha arcoíris. Otra ST, ST45, contiene cepas de Chile, Estados Unidos y Canadá (British Columbia), todas provenientes de salmón Coho. El mayor número de cepas, 48, fue obtenido de órganos internos, como riñón, bazo, hígado y gónadas, mientras que 38 se aislaron de tejidos superficiales como branquias, aletas y ulceras en la piel. Los resultados de MLST no permiten realizar una clara separación entre cepas sistémicas y aquellas obtenidas de tejidos superficiales.
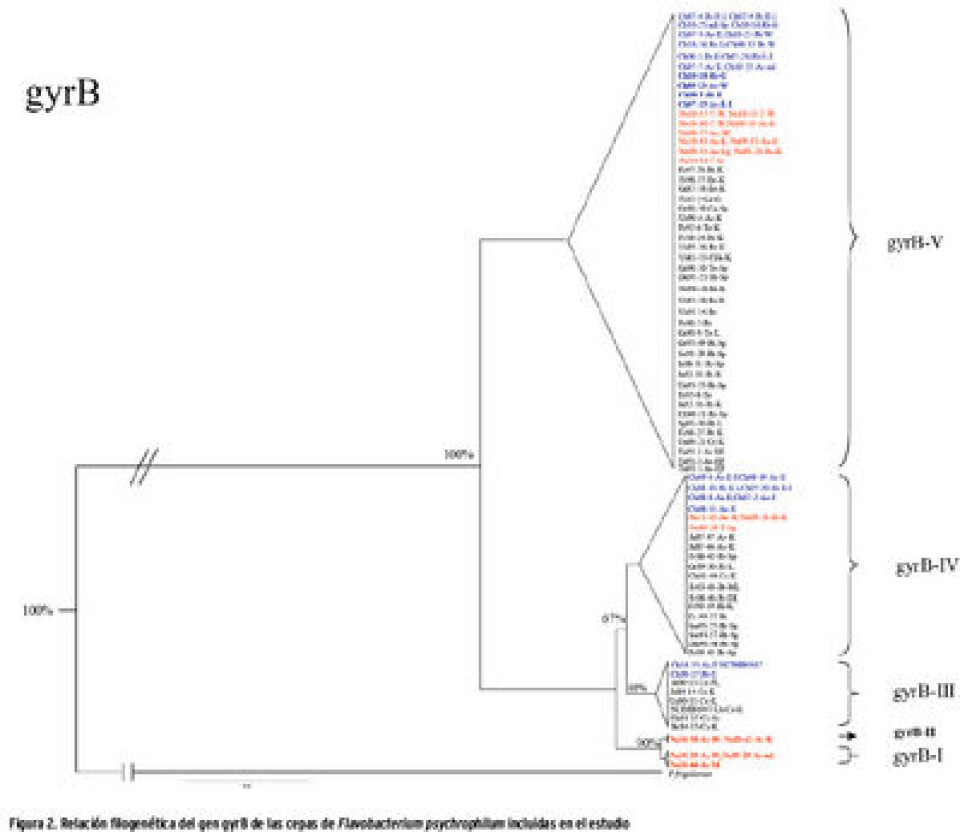
Discusión Se ha descrito ampliamente en la literatura la importancia de realizar la caracterización genética de F. Psychrophilum, con el objetivo de definir marcadores de virulencia que permitan diferenciar las cepas en relación a los mecanismos involucrados en la patogénesis, como base para el desarrollo de vacunas, para el control y tratamiento de la flavobacteriosis, y para encontrar marcadores genéticos que permitan la trazabilidad de las cepas durante un brote de la enfermedad, para así definir patrones de transmisión. Para F. psychrophilum no se ha definido hasta ahora un método estándar de caracterización genética, sin embargo, Nicolas et al. (2008), describe un promisorio método para entender la estructura como población de este patógeno. La amplificación y secuenciación del gen 16S rRNA se usa comúnmente para la detección e identificación de patógenos bacterianos en peces incluyendo miembros del género Flavobacterium, sin embargo, este estudio demuestra que la resolución entregada por las secuencias nucleotídicas de este gen no es suficiente para separar las diferentes cepas de la bacteria. En relación con la filogenia de los siete HG, cada uno de esos genes, analizados separadamente, entregaron una información limitada de la relaciones filogenéticas entre las cepas. La mejor resolución la entrego gyrB con cinco clades (Fig. 2). En éstas se aprecia que algunas cepas de la misma especie noruega de salmónidos en gyrB-I and gyrB-II se agruparon cercanas filogenéticamente. En MLST (Figura 3), se determinaron 53 STs entre las 44 cepas de Chile y Noruega y la mayoría se obtuvieron de trucha arcoíris (N = 39) y salmón del Atlántico (N = 27), considerando los 50 cepas de Nicolas et al. (2008). Hasta ahora, no existe claridad en relación al rango de reservorios naturales de F. psychrophilum, incluidas las especies no-salmónidas, en relación a su rol en la transmisión del patógeno hacia los salmónidos, aunque a través de las cepas disponibles se podrían extraer algunas conclusiones. Las dos cepas noruegas en ST 6 provienen de sitios de trucha arco iris y de salmón del Atlántico de cultivo del mismo fiordo y de la misma empresa. Los dos aislados probablemente arribaron a los sitios cuando los smolts fueron puestos en el mar. Sin embargo, debido a las lluvias, las aguas del fiordo pudieron haber disminuido su salinidad permitiendo la transmisión horizontal de F. psychrophilum entre los sitios, aunque las cepas No09-29As, No10-42As (ST49) y No10-44As (ST48) fueron obtenidas en el mismo periodo de tiempo en el mismo fiordo de salmónidos nativos migrando por el río al fiordo, registrándose diferentes STs. En la ST42, las cinco cepas provienen de salmón del Atlántico y truchas silvestres del mismo río en el noroeste de Noruega, lo que sugiere que la transmisión horizontal inter-especie de cepas puede ser frecuente, ya que en este río conviven sólo estas dos especies. Así, los resultados de este estudio no confirman la especificidad por un determinado hospedador en Noruega. Tampoco se puede confirmar lo mismo entre las cepas chilenas, ya que ST9, ST16 y ST20 contiene varias cepas de F. psychrophilum obtenidas desde salmón del Atlántico y trucha arcoíris. Es esperable que las poblaciones de patógenos de peces deberían ser influenciadas por acuicultura intensiva, lo que permitiría la combinación de cepas que pudieran haber tenido previamente un hospedador específico. Analizar un número mayor de cepas obtenidas de diferentes especies, incluidas las de no-salmónidos, pudiera clarificar un posible patrón relacionado con hospedador específico, como ha sido sugerido por otros autores (Arai et al., 2007, Kim et al., 2010). En Chile, todas las especies de salmónidos has sido introducidas desde Norteamérica y Europa. La primera introducción ocurrió el año 1905, con la importación de 400.000 ovas de salmón del Atlántico y trucha arcoíris desde Alemania (Avila et al., 1994). Otras importaciones has provenido desde Estados Unidos, Irlanda, Dinamarca, Escocia, Suecia y Noruega. Junto al movimiento de ovas también está el riesgo de introducir organismos, parásitos, virus o bacterias en áreas libres de estos (Vike et al., 2009). De acuerdo con la alta prevalencia de F. psychrophilum en la acuicultura chilena, ésta puede ser resultado de la introducción de la bacteria a través de la importación de ovas (Bustos et al., 1995). Hasta ahora no se ha aislado F. psychrophilum desde peces nativos en Chile por lo que no hay reportes de flavobacteriosis en ellas. Sin embargo, la ausencia de reportes no implica necesariamente la ausencia de F. psychrophilum en especies nativas en Chile, pero si las cepas chilenas del patógeno pudieran haber sido introducidas, entonces deberían estar estrechamente relacionadas en sus características genéticas con las de Europa y Norteamérica. En el dendrograma MLST, todas las cepas chilenas están distribuidas entre las de Europa y Norteamérica, indicando que los salmónidos de cultivo en Chile no se han infectado con cepas autóctonas de F. psychrophilum. Así, no se confirma la hipótesis de la existencia de cepas geográficamente distintas entre las cepas de Noruega y Chile.
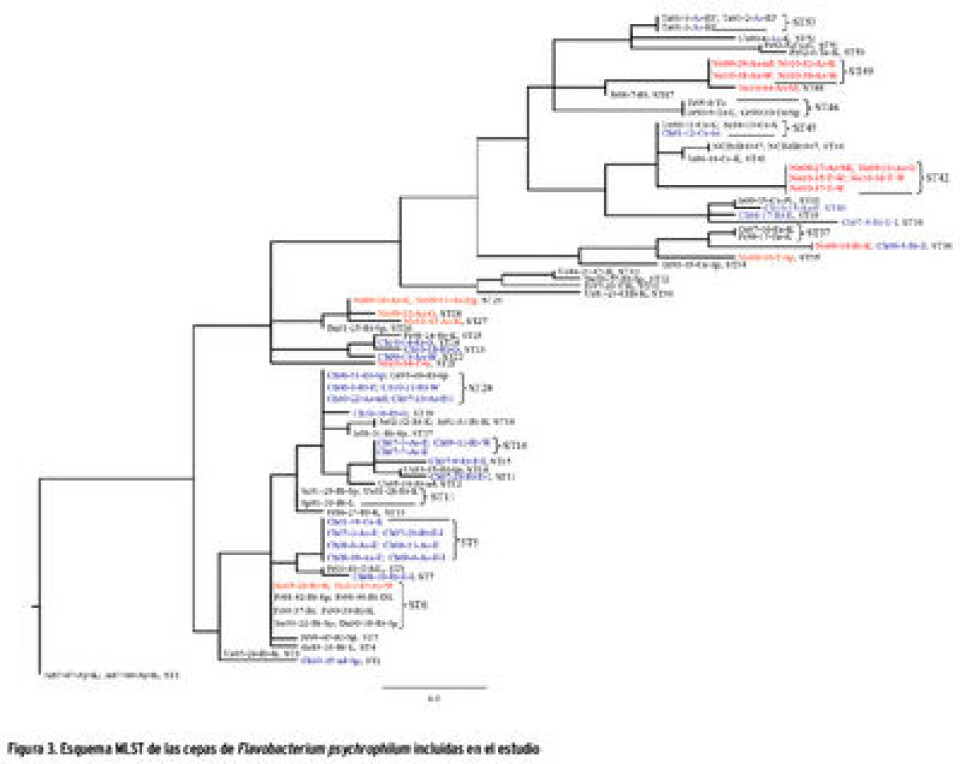
Relacionado con tropismo por tejidos de F. psychrophilum, éste se ha aislado desde branquias, piel, aletas y heridas, entre otros. El pez puede mostrar signos de la enfermedad o presentar diversas ulceras en la piel y aletas o estar moribundo teniendo seriamente afectadas las branquias o presentar serias hemorragias en la musculatura, lo que sugiere una amplia variación en la virulencia entre cepas del patógeno. En este estudio F. psychrophilum fue aislado desde peces presumiblemente sanos hasta moribundos con serio compromiso de branquias, extendidas y profundas úlceras en la cabeza, líneas laterales y dorsal y pedúnculo. También fue obtenido desde gónadas de salmón del Atlántico al desove, sin embargo, no se pudo establecer un patrón en los análisis de MLST. De esta forma, el presente esquema MLST no identifica potenciales cepas patogénicas/ virulentas de F. psychrophilum.
Referencias Arai, H., et al. (2007) Molecular typing by pulsed-field gel electrophoresis of Flavobacterium psychrophilum isolates derived from Japanese fish. Journal of Fish Diseases, 30, 345-355. Avila, M., et al. (1994) Status and perspectives of aquaculture in Chile. Corfo-Ifop 166 . (in Spanish). Borg, A.F. (1948) Studies on myxobacteria associated with diseases in salmonid fishes. PhD thesis, University of Washington. Brevik, O. J., Ottem, K. F. & Nylund, A. (2011) Multiple-locus, variable number of tandem repeat analysis (MLVA) of the fish-pathogen Francisella noatunensis. Bmc Veterinary Research, 7. Bustos, P.A., et al. (1995) First isolation of Flexibacter psychrophilus, as causative agent of rainbow trout fry syndrome (RFTS), producing rainbow trout mortality in Chile. Bulletin of the European Association of Fish Pathologists, 15, 162-163. Cipriano, R. C. & Holt, R. A. (2005) Flavobacterium psychrophilum, cause of Bacterial Cold-Water Disease and Rainbow Trout Syndrome. Fish Disease Leaflet No. 86. United States Dept of the interior. U.S. Geological Service, National Fish Health Research Laboratory, Kearneyville, WV. Duesund, H., et al. (2010) Characterization of a VHS virus genotype III isolated from rainbow trout (Oncorhychus mykiss) at a marine site on the west coast of Norway. Virology Journal, 7. Hesami, S., et al. (2008) Phenotypic and genotypic analysis of Flavobacterium psychrophilum isolates from Ontario salmonids with bacterial coldwater disease. Canadian Journal of Microbiology, 54, 619-629. Kim, J. H., et al. (2010) Plasmid profiling of Flavobacterium psychrophilum isolates from ayu (Plecoglossus altivelis altivelis) and other fish species in Japan. Journal of Veterinary Science, 11, 85-87. Kumagai, A. & Takahashi, K. (1997) Imported eggs responsible for the outbreaks of cold-water disease among cultured Coho salmon in Japan. Fish Pathology, 32, 231-232. Madetoja, J., Nyman, P. & Wiklund, T. (2000) Flavobacterium psychrophilum, invasion into and shedding by rainbow trout Oncorhynchus mykiss. Diseases of Aquatic Organisms, 43, 27-38. Nicolas, P., et al. (2008) Population structure of the fish-pathogenic bacterium Flavobacterium psychrophilum. Applied and Environmental Microbiology, 74, 3702-3709. Nilsen, H., et al. (2011) Flavobacterium psychrophilum associated with septicaemia and necrotic myositis in Atlantic salmon Salmo salar: a case report. Diseases of Aquatic Organisms, 97, 37-46. van Belkum, A., et al. (2007) Guidelines for the validation and application of typing methods for use in bacterial epidemiology. Clinical Microbiology and Infection, 13, 1-46. Vike, S., Nylund, S. & Nylund, A. (2009) ISA virus in Chile: evidence of vertical transmission. Archives of Virology, 154, 1-8.
















