Científicos asocian a Tenacibaculum y Dokdonia con Caligus
Chile: Como parte de la microbiota del piojo de mar, la interacción con estas bacterias podría ayudar a desarrollar nuevas estrategias de control en salmonicultura, según un nuevo estudio.
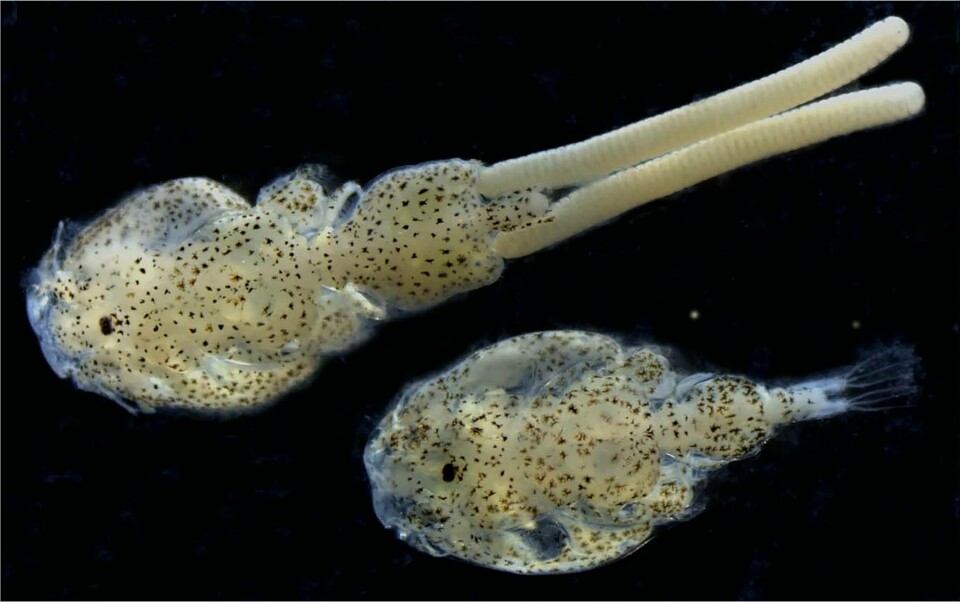
La microbiota asociada a organismos puede desempeñar funciones clave en el desarrollo de casi todos los organismos vivos. En los parásitos, esta función puede comprender tanto funciones biológicas como funcionales durante la patogénesis.
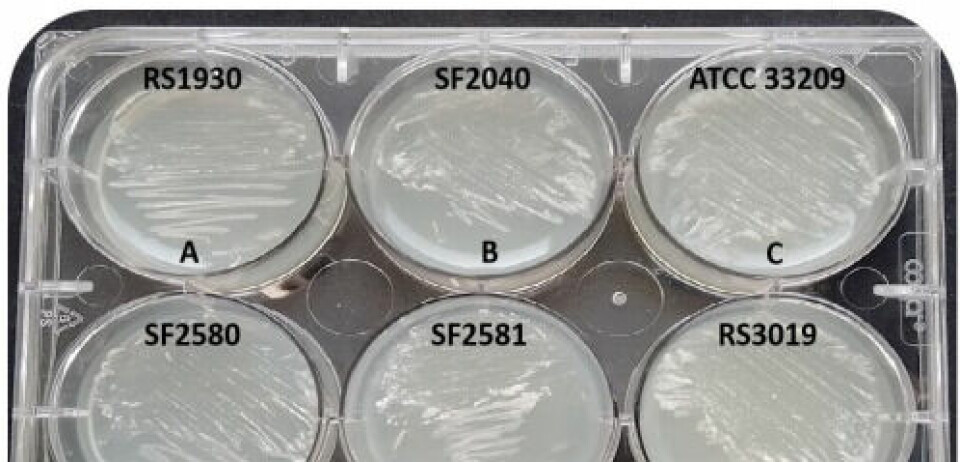
El Dr. Diego Valenzuela, investigador del Centro Incar, ha sido un de los expertos en estudiar justamente el rol de la microbiota asociada a Caligus, identificando 413 grupos de genomas bacterianos, incluidos seis genomas bacterianos con más del 80% de integridad.
El genoma bacteriano más representado perteneció la bacteria Tenacibacullum ovolyticum (completitud del 97,87%), seguido de Dokdonia sp. (96,71% de compleción).
Estos resultados, junto con un estudio más a fondo de los mismos, fueron publicados recientemente, donde en asociación con otros científicos del Centro Incar, la Universidad de Algarve y Phase Genomics explicaron a fondo la significancia de estos resultados.
Tenacibaculum
Con la secuenciación del gen 16S, los expertos encontraron cierta relación con las bacterias patógenas T. maritimum y T. dicentrarchi. En base a esto, luego buscaron posibles factores de virulencia, identificando un total de 21 genes presentes en el genoma de T. ovolyticum, incluidos los asociados al sistema de secreción Dot/Icm tipo IVB, proteínas de choque térmico (HtpB) y genes de catalasas (katA), entre otros.
“Además, utilizando la base de datos MEGARes, los resultados evidenciaron genes de resistencia a los antibióticos (ARG) dentro de todos los grupos del genoma bacteriano. Fue posible identificar cuatro ARG, incluido el transportador de eflujo de tetraciclina Na+/H+ tet, la proteína de resistencia a quinolonas mediada por plásmidos (QnrS2) , la cloranfenicol acetiltransferasa de Integron (catB9) y la oxa betalactamasa (Oxa-209)”, informaron los autores.
Dokdonia
En cuanto al genoma de la otra bacteria identificada, pudieron evidenciar la presencia de genes asociados con diferentes características del metabolismo, incluido el metabolismo de proteínas, aminoácidos y derivados, seguidos de cofactores, vitaminas, pigmentos y otros.
No obstante, la mayoría de los genes se relacionaron a la biosíntesis, degradación, procesamiento y modificación de proteínas.
“Los resultados evidenciaron que el mayor número de genes metabólicos anotados en la microbiota de Caligus, pero no presentes en el genoma del piojo de mar, pertenecen al metabolismo de aminoácidos, seguido del metabolismo de carbohidratos y lípidos”, explicaron los investigadores.
Aunque no pudieron confirmar la complementación metabólica entre el piojos de mar y su microbiota, los expertos plantearon que estos resultados “podrían sugerir nuevos conocimientos sobre los posibles roles de la microbiota asociada a los piojos de mar en la biología de los parásitos”.
“Creemos que nuestros hallazgos deberían generar conciencia sobre la microbiota del piojos de mar como un componente fundamental en la acuicultura del salmón, proporcionando nuevas perspectivas para desarrollar nuevas estrategias de control”, concluyeron los especialistas.
Lea el estudio completo titulado “Proximity ligation strategy for the genomic reconstruction of microbial communities associated with the ectoparasite Caligus rogercresseyi“, aquí.