Avances en el mejoramiento genético de la resistencia al Síndrome Rickettsial del Salmón (Pisciricketsia salmonis): integrando la información del genoma
José M. Yáñez1,2,*, Agustín Barría1,3, Katharina Correa1,2, Liane Bassini3, Jean P. Lhorente2 1 Facultad de Ciencias Veterinarias y Pecuarias, Universidad de Chile, Av Santa Rosa 11735, La Pintana, Santiago, Chile 2 Aquainnovo, Talca 60, Puerto Montt, Chile 3 Facultad de Ciencias Agronómicas, Universidad de Chile, Av Santa Rosa 11315, La Pintana, Santiago, Chile * Correspondencia: José M. Yáñez, jmayanez@uchile.cl www.favet-genstat.uchile.cl
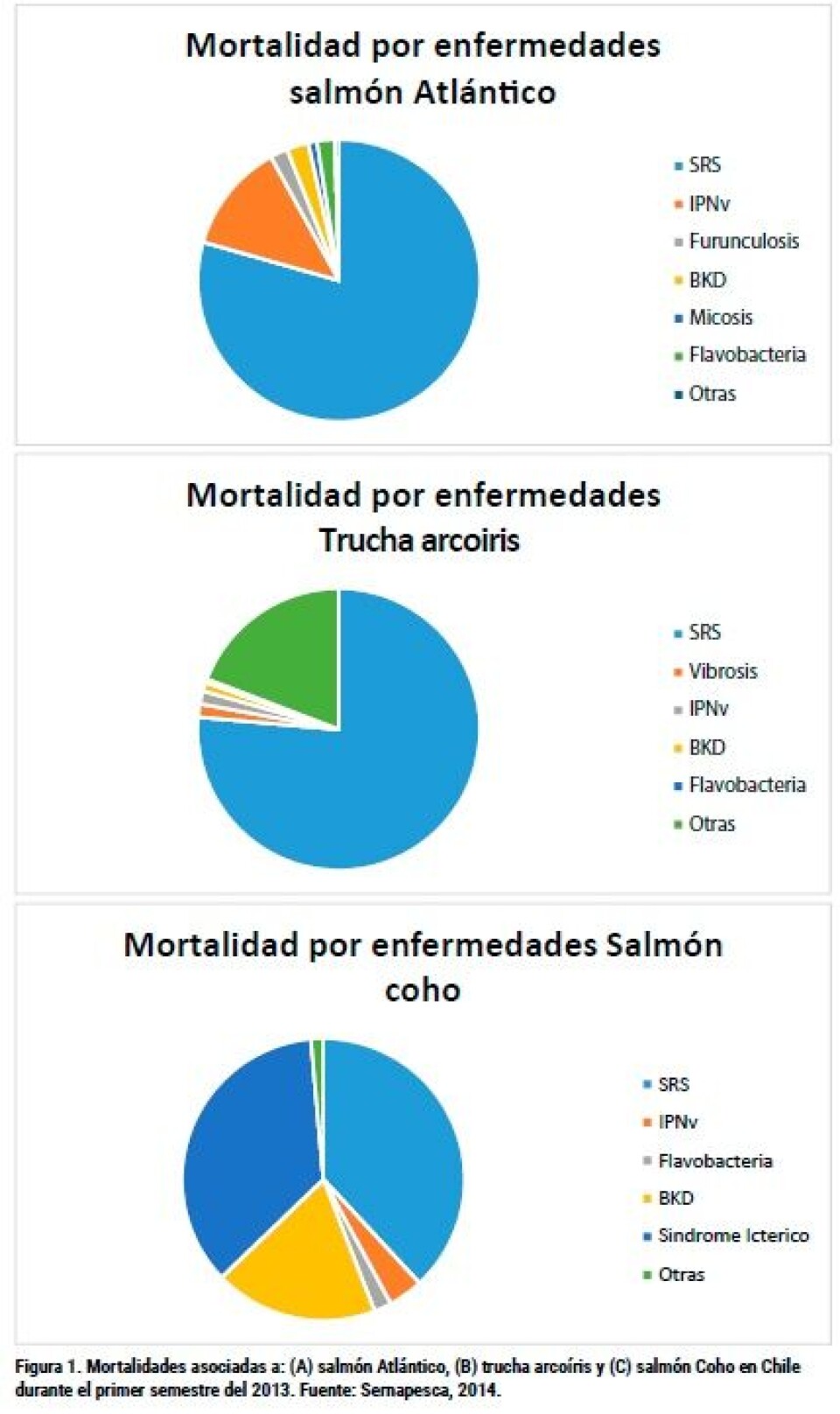
Introducción El estado sanitario de los peces cultivados es uno de los principales desafíos para la industria salmonicultora, tanto en nuestro país como a nivel global, siendo los brotes de enfermedades uno de los principales factores que afectan el retorno económico de este sector productivo. En Chile, uno de los mayores problemas sanitarios que afectan el cultivo de especies salmónidas es el Síndrome Rickettsial del Salmón (SRS), enfermedad infecciosa cuyo agente etiológico es una bacteria gram-negativa intracelular facultativa denominada Piscirickettsia salmonis, la cual fue aislada inicialmente a partir de una especie de salmón del Pacífico en nuestro país (Cvitanich y col., 1991). Esta enfermedad es capaz de afectar a las tres principales especies salmonideas producidas en nuestro país: salmón Atlántico (Salmo salar), trucha arcoíris (Oncorhynchus mykiss) y salmón Coho (Oncorhynchus kisutch) (Fryer y Hedrick, 2003), siendo responsable de más de la mitad del total de las mortalidades asociadas con enfermedades infecciosas (Sernapesca, 2014). Durante el primer semestre del 2014, SRS ocasionó el 79%, 76% y 38% de las mortalidades asociadas con causas infecciosas en salmón Atlántico, trucha arcoíris y salmón Coho, respectivamente (Figura 1) (Sernapesca, 2014). Las distintas estrategias implementadas para la prevención y control de SRS no han presentado los resultados esperados en términos de efectividad. Por ejemplo, a pesar que algunas vacunas han mostrado resultados positivos en desafíos experimentales, a la fecha no han sido capaces de demostrar la misma efectividad en la reducción de la mortalidad en condiciones de cultivo (Rozas y Enríquez, 2014). En tanto, se ha descrito que el uso de antibióticos posee un efecto limitado sobre la enfermedad durante los brotes (Cabello, 2006; Rozas y Enríquez, 2014). Se ha sugerido que estos resultados pueden estar asociados con la dificultad por parte de las drogas para lograr una concentración intracelular adecuada (Fryer y Hedrick, 2003). Además, se han reportado cepas de P. salmonis resistentes a ciertos antibióticos. Adicionalmente, durante el 2013, el 92% del total de los antibióticos utilizados en la industria salmonicultora, en fase de cultivo de mar, fueron administrados para el control de SRS (Sernapesca, 2013), lo cual constituye además, un problema de salud pública y medio ambiental. Es por esto que resulta imperativo desarrollar nuevas estrategias para el control y prevención de esta enfermedad. En este sentido, se ha propuesto que el mejoramiento genético de la resistencia a enfermedades puede constituir una estrategia factible y sustentable para la prevención y control, permitiendo además reducir la utilización de antibióticos, contribuir al bienestar animal y a un menor impacto sobre el medio ambiente (Bishop y Woolliams, 2014).
Mejoramiento genético en salmónidos Los programas de mejoramiento genético han permitido incrementar el retorno económico de las explotaciones agropecuarias. El objetivo de mejoramiento debe ser definido para cada especie y población. En este contexto, todo carácter heredable y de relevancia económica debe ser incluido dentro de este objetivo, como lo son tasa de crecimiento, color de carne y resistencia a enfermedades en salmónidos (Gjedrem, 2012). Chile ha sido pionero en el mejoramiento genético de especies salmónidas, estableciendo en 1992 el primer programa de selección artificial para crecimiento corporal en salmón Coho (Neira y col., 2006). Adicionalmente, durante los años 1997 y 2000 se establecieron los primeros programas comerciales de mejoramiento genético para salmón Atlántico y trucha arcoíris respectivamente (Neira, 2010).
Resistencia genética a enfermedades La resistencia a enfermedades puede definirse como la habilidad del huésped para limitar la infección mediante la reducción en la tasa de replicación del patógeno (Doeschl-Wilson y col., 2012). El seleccionar animales con una mayor resistencia para una enfermedad específica es un método factible para mejorar la productividad y el bienestar animal, además de poseer ventajas sobre otros métodos de control de enfermedades, como los beneficios permanentes y acumulativos otorgados por el mejoramiento genético (Bishop y Woolliams, 2014). Típicamente, la mortalidad inducida por la infección es utilizada para definir el rasgo de resistencia a enfermedades en peces (Ødegård y col., 2011, Yáñez y col., 2014a). El mejoramiento genético de la resistencia a enfermedades apunta a seleccionar animales que tengan una respuesta inmune eficiente frente a una infección determinada, manifestándose ésta a través de una mayor sobrevivencia. Los registros de sobrevivencia que suelen ser utilizados en la evaluación de la resistencia genética a enfermedades en peces provienen, generalmente, de brotes de campo y desafíos experimentales controlados (Yáñez y col., 2014a). Los desafíos experimentales controlados son más apropiados que las pruebas de campo, ya que permiten manejar las condiciones ambientales y lograr obtener heredabilidades de mayor magnitud (Ødegård et al., 2011). En los desafíos experimentales, peces con genealogía conocida y marcados individualmente son expuestos al patógeno, con inóculos mediante inmersión, cohabitación o inyección intraperitoneal (IP), con el objetivo de registrar las curvas de mortalidad (Nordmo y col.,y col., 1997).
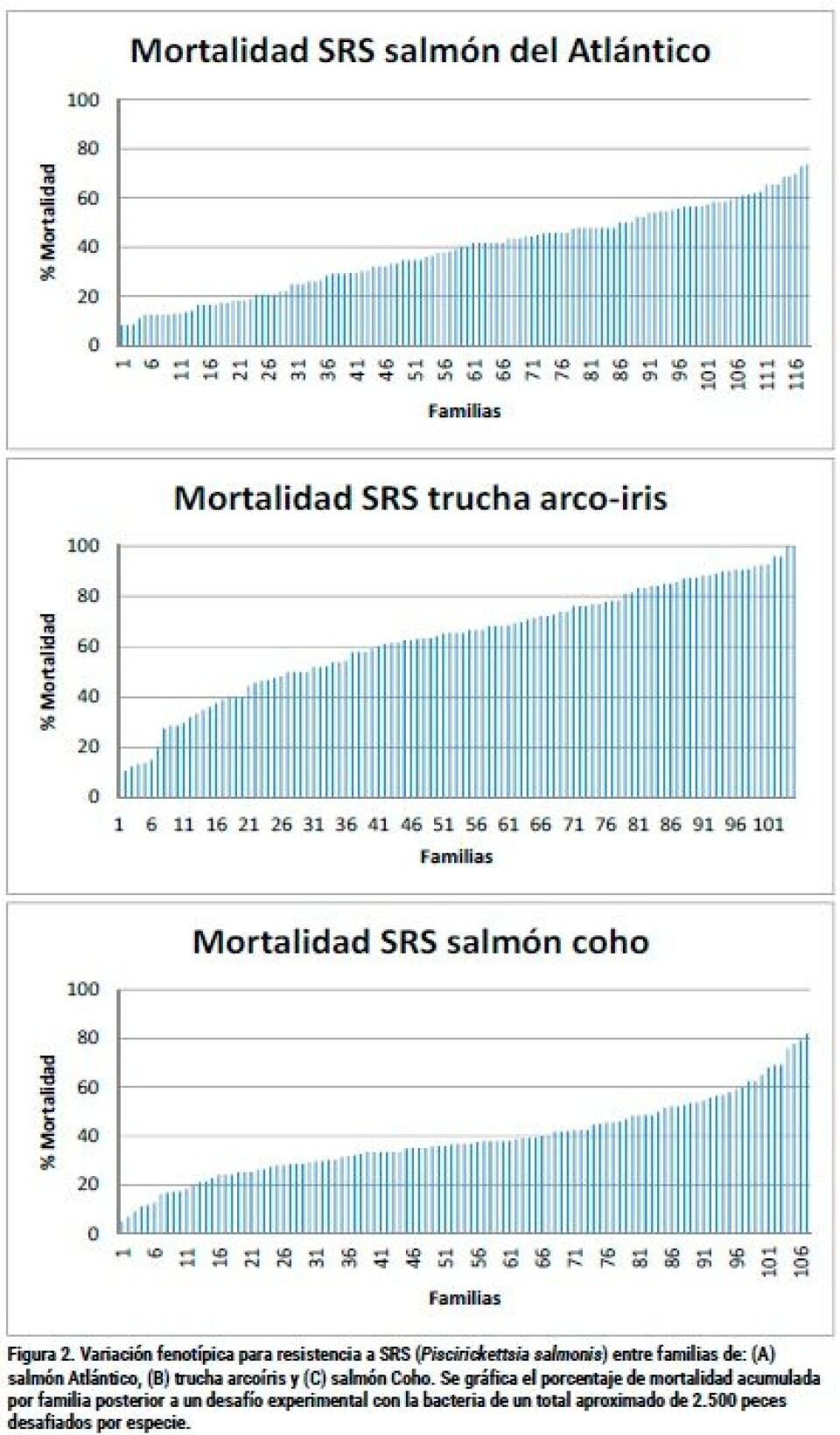
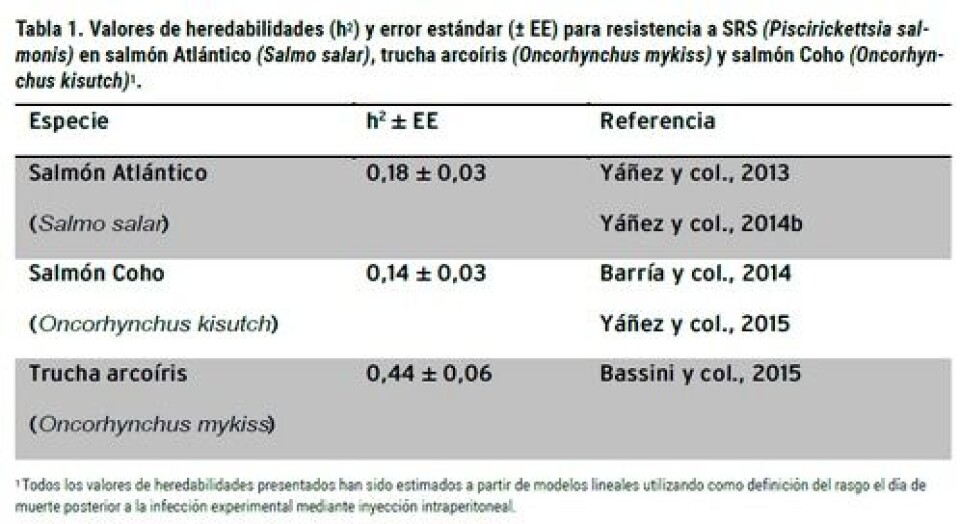
Un requisito necesario para el mejoramiento de un rasgo mediante selección artificial es la existencia de variación genética para ese carácter dentro de la población. Dentro de este contexto, la heredabilidad es la proporción de la varianza fenotípica total atribuida a la variación genético-aditiva (Falconer y Mackay, 1996). Actualmente, diversos estudios realizados por la Unidad de Genética y Genómica Acuícola (Facultad de Ciencias Veterinarias y Pecuarias, Universidad de Chile) y Aquainnovo, han demostrado niveles de variación fenotípica y genética significativa para la resistencia a SRS en salmón Atlántico (Yáñez et al., 2014b, 2013), salmón Coho (Barría y col., 2014; Yáñez y col., 2015) y trucha arcoíris (Bassini y col., 2015). La Figura 2 y la Tabla 1, muestran la variación fenotípica para la resistencia a SRS y los valores de heredabilidad para el mismo rasgo en las tres especies, respectivamente. Estos resultados demuestran la factibilidad de mejorar el carácter de resistencia a SRS mediante el mejoramiento genético en salmónidos, lo cual constituye una alternativa para el control de esta enfermedad. Además, se han estimado correlaciones genéticas entre resistencia a SRS y peso corporal en las tres especies, las cuales van desde cero en trucha arcoíris (Bassini y col., 2015) hasta valores negativos en salmón del Atlántico (-0,19 ± 0,12) (Yáñez y col., 2014b) y salmón Coho (-0,44 ± 0,12) (Yáñez y col., 2015). A su vez, se han estimado correlaciones genéticas entre resistencia a SRS y resistencia al parásito Caligus rogercresseyi en trucha arcoíris y salmón Atlántico, las cuales en ambos casos no han sido significativamente distintas de cero (-0,07 ± 0,14 y -0,02 ± 0,17, respectivamente) (Bassini y col., 2015; Yáñez y col., 2013b). Estas correlaciones genéticas deben ser tomadas en consideración al momento de incorporar estos rasgos de forma simultánea dentro del objetivo de mejoramiento. A la fecha, los programas de mejoramiento en salmones han incluido la resistencia a enfermedades utilizando la información de parientes, situación que afecta el progreso genético alcanzable en cada generación, debido a una reducida precisión de los valores genéticos estimados (Estimated Breeding Values o EBVs), en comparación a la precisión obtenida al utilizar la información de los propios candidatos a la selección.
Incorporación de información genómica El desarrollo e implementación de nuevas tecnologías aplicadas al estudio de genomas completos, tales como métodos de secuenciación masiva y plataformas de genotipificación de alto rendimiento, en conjunto con la disminución de los costos asociados, han generado un rápido incremento en la disponibilidad de recursos genómicos en especies salmónidas y otros peces de cultivo (Yáñez y col., 2015b). Estas nuevas tecnologías han permitido generar importantes herramientas para la identificación de las variantes genéticas y genes involucrados en los caracteres de resistencia a enfermedades. Chile ha formado parte de la International Collaboration to Sequence the Atlantic Salmon Genome (Icsasg), cuyo objetivo ha sido secuenciar el genoma y generar recursos genómicos para el salmón Atlántico (Davidson y col., 2010). La última versión del genoma de esta especie se encuentra disponible públicamente desde el 22 de abril del 2015 (http://www.ncbi.nlm.nih.gov/nuccore/AGKD00000000). En tanto, también se ha liberado una versión del genoma de trucha arcoíris (Berthelot y col., 2014). Junto con esto, y en colaboración con investigadores canadienses, nuestro grupo de investigación participará activamente en el proyecto de secuenciación del genoma de salmón Coho, el cual será financiado por entidades públicas y privadas, tanto de Chile como de Canadá (Davidson, 2015, comunicación personal). Estos avances han permitido el desarrollo de plataformas genotipificación de marcadores moleculares del tipo SNP (Single Nucleotide Polymorphisms) de alta densidad para salmón Atlántico y trucha arcoíris (Palti y col., 2014; Yañez y col., 2014). Estos recursos genómicos pueden ser utilizados para la identificación de las variantes genéticas y genes involucrados en rasgos complejos (por ejemplo, resistencia a SRS) en estas especies, además de permitir su incorporación en programas de mejoramiento. La información obtenida a partir de unos pocos marcadores moleculares ligados a un Locus de Efecto Cuantitativo (Quantitative Trait Locus o QTL), podría ser implementada en programas de mejoramiento mediante selección asistida por marcadores (MAS), en el caso de que sean capaces de explicar una proporción importante de la variación genética para el rasgo. No obstante, la información de una gran cantidad de marcadores moleculares debería ser incluida, en lo que se ha denominado selección genómica (GS), si la variación explicada por cada uno de estos marcadores es pequeña (Meuwissen y col., 2001). Estas metodologías basadas en la incorporación de información molecular poseen un elevado potencial para acelerar el mejoramiento de la resistencia a enfermedades, debido que permitirán predecir el mérito genético de los reproductores con mayor precisión que al utilizar solamente la información de genealogías y fenotipos (Taylor, 2014; Yáñez y col., 2014).No obstante, para poder aplicar esquemas de selección que incorporen información molecular, ya sea MAS o GS, en primer lugar se deben conocer los factores genéticos que explican la variación cuantitativa para una determinada característica. Estos factores genéticos pueden ser identificados mediante metodologías genético-estadísticas que explotan la información combinada proveniente de registros fenotípicos y marcadores moleculares SNPs de alta resolución, en lo que se ha denominado estudios de asociación genómica (Genome Wide Association Studies o GWAs) (Goddard y Hayes, 2009).
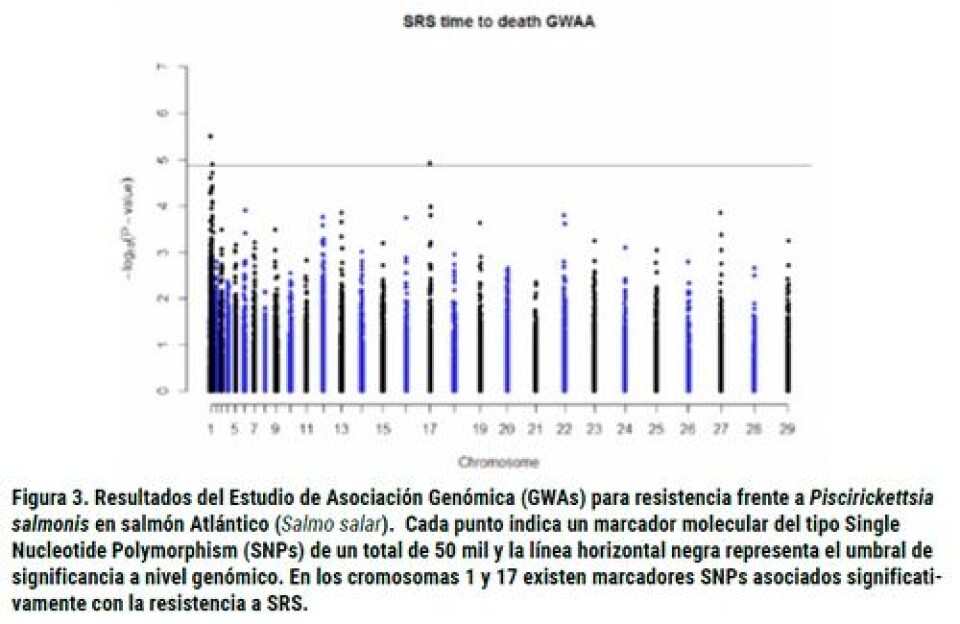
De esta forma, mediante GWAs hemos descubierto las regiones genómicas o QTLs relacionados con la resistencia a SRS en salmón Atlántico. La Figura 3 muestra los resultados de GWAs para el carácter de resistencia a SRS definido como día de muerte posterior a la infección experimental y 50 mil marcadores moleculares tipo SNPs distribuidos de manera uniforme a través del genoma. Este gráfico demuestra que los cromosomas 1 y 17 de salmón Atlántico albergan variantes genéticas o genes asociados de forma significativa a la resistencia frente a SRS (puntos que sobrepasan la línea negra horizontal, la cual indica el umbral de significancia) (Correa y col., 2015). Sin embargo, el porcentaje de la variación total de la resistencia explicada por estos QTLs es reducida (menor al 10% del total). Estos resultados sugieren que, al menos para salmón Atlántico, serán necesarios marcadores moleculares distribuidos a través de todo el genoma para poder capturar el efecto de todos los QTLs de pequeña magnitud involucrados en la resistencia a SRS y, de esta forma, incrementar la precisión en las evaluaciones genéticas del rasgo mediante una aproximación de GS.
Direcciones futuras Los avances en la secuenciación del genoma de especies salmónidas, en conjunto con la disponibilidad de estrategias de secuenciación masiva y paneles de genotipificación de SNPs de alto rendimiento para estas especies, representan una oportunidad única para el descubrimiento de nuevas variantes genéticas involucradas en la resistencia a SRS con una mayor resolución. Adicionalmente, estas nuevas estrategias, aplicadas a las tres principales especies salmónidas producidas en Chile mediante un enfoque integrador, ayudarán en la comprensión de los mecanismos genéticos asociados con la resistencia a SRS. Esto último permitirá la identificación de regiones genómicas acotadas o genes involucrados en la respuesta inmune diferencial frente a esta bacteria. Actualmente, nuestro grupo de investigación, en la Facultad de Ciencias Veterinarias y Pecuarias de la Universidad de Chile, en conjunto con The Roslin Institute - The University of Edinburgh (Escocia) se encuentra trabajando en una aproximación integrativa para el mejoramiento genético de la resistencia a SRS, utilizando información del genoma de las tres especies, gracias a la adjudicación del proyecto: “Development of an integrative strategy for the genetic improvement of the resistance against Salmon Rickettsial Syndrome (Piscirickettsia salmonis) in salmonid species using molecular information”, financiado por el fondo Newton-Picarte (Fondef-Conicyt y The British Council). La disponibilidad de esta información permitirá el desarrollo de nuevas alternativas para el control de SRS mediante la selección genética de reproductores más resistentes con una elevada eficiencia. De esta forma, se apuntará a disminuir los niveles de mortalidad asociados con SRS, utilizando una biotecnología sustentable y amable con el medioambiente.
Agradecimientos Agustín Barría y Katharina Correa agradecen a Becas de Doctorado CONICYT. Liane Bassini agradece a Becas de Doctorado Capes (Gobierno de Brasil). Este trabajo ha sido financiado en parte por los proyectos Innova-Chile de Corfo (11IEI-12843), Newton-Picarte: Fondef-Conicyt y The British Council (IT14I10100) y U-inicia, VID, Universidad de Chile.
Referencias Barría A, Duffloqc P, Lhorente JP, Oyarzún M, Yáñez JM (2014) Resistencia genética a Piscirickettsia salmonis en salmon coho (Oncorhynchus kisutch). V Congreso de Acuicultura. Coquimbo, Chile. Bassini LN, Neira R, Yáñez JM, Newman S, Oyarzún M, Erranz F, Figueroa R, Lhorente JP (2015) Genetic parameters for resistance to Caligus rogercresseyi, Piscirickettsia salmonis and body weight in rainbow trout (Oncorhynchus mykiss). The International Symposium on Genetics in Aquaculture XII. Santiago de Compostela, España. Berthelot C, Brunet F, Chalopin D, Juanchich A, Bernard M, Noël B, Bento P, Da Silva C, et al (2014) The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates. Nat. Commun. 5, 3657. Bishop SC, Woolliams JA (2014) Genomics and disease resistance studies in livestock. Livestock Science, 166, 190-198. Cabello FC (2006) Heavy use of prophylactic antibiotics in aquaculture: a growing problem for human and animal health and for the environment. Environ. Microbiol. 8, 1137–1144. Correa K, Lhorente JP, López ME., Bassini L, Di Genova A, Maass A, Davidson W, Yáñez JM (2015) Genome wide association analysis reveals genetic architecture of the resistance to Piscirickettsia salmonis in Atlantic salmon (Salmo salar L.). The International Symposium on Genetics in Aquaculture XII. Santiago de Compostela, España. Cvitanich J, Gárate O, Smith CE (1991) The isolation of a rickettsia like organism causing disease and mortality in Chilean salmonids and its confirmation by Koch’s postulate. J. Fish Dis. 14, 121–146. Davidson W, Koop B, Jones S, Iturra P, Vidal R, Maass A, Jonassen I, Lien S, Omholt S (2010) Sequencing the genome of the Atlantic salmon (Salmo salar). Genome Biol. 11, 403. Doeschl-Wilson AB, Bishop SC, Kyriazakis I, Villanueva B (2012) Novel methods for quantifying individual host response to infectious pathogens for genetic analyses. Front. Genet. 3, 1–9. Falconer DS, Mackay TFC (1996) Introduction to Quantitative Genetics. Fryer JL, Hedrick, RP (2003) Piscirickettsia salmonis: a Gram-negative intracellular bacterial pathogen of fish. J. Fish Dis. 247, 251–262. Gjedrem T (2012) Genetic improvement for the development of efficient global aquaculture: A personal opinion review. Aquaculture 344-349, 12–22. Goddard ME, Hayes BJ (2009) Mapping genes for complex traits in domestic animals and their use in breeding programmes. Nat. Rev. Genet. 10, 381–391. Meuwissen TH, Hayes BJ, Goddard ME (2001) Prediction of Total Genetic Value Using Genome-Wide Dense Marker Maps. Genetics 157, 1819–1829. Neira, R, 2010. Breeding in aquaculture species: genetic improvement programs in developing countries, in: 9th Wcgalp. Leipzig, Alemania. Neira R, Díaz NF, Gall G, Gallardo J, Lhorente JP, Alert A (2006) Genetic improvement in coho salmon (Oncorhynchus kisutch). II: Selection response for early spawning date. Aquaculture 257, 1–8. doi:10.1016/j.aquaculture.2006.03.001 Palti Y, Gao G, Liu S, Kent MP, Lien S, Miller MR, Rexroad CE, Moen T (2014) The development and characterization of a 57K single nucleotide polymorphism array for rainbow trout. Mol. Ecol. Resour. (In Press) Rozas M, Enríquez R (2014) Piscirickettsiosis and Piscirickettsia salmonis in fish: a review. J. Fish Dis. 37, 163–188. Sernapesca, Informe Sobre uso de Antimicrobianos en la Salmonicultura Nacional (2013). Sernapesca, Situación Sanitaria Salmonicultura Centros Marinos (2014). Taylor J, (2014) Implementation and accuracy of genomic selection. Aquaculture 420-421, S8–S14. Yáñez JM, Bangera R, Lhorente JP, Oyarzún M, Neira R (2013) Quantitative genetic variation of resistance against Piscirickettsia salmonis in Atlantic salmon (Salmo salar). Aquaculture 414-415, 155–159. Yáñez JM, Houston RD, Newman S (2014a). Genetics and genomics of disease resistance in salmonid species. Frontiers in genetics, 5. Yáñez JM, Newman S, Houston RD (2015). Genomics in aquaculture to better understand species biology and accelerate genetic progress. Frontiers in genetics, 6. Yáñez JM, Lhorente JP, Bassini LN, Oyarzún M, Neira R, Newman S (2014b) Genetic co-variation between resistance against both Caligus rogercresseyi and Piscirickettsia salmonis, and body weight in Atlantic salmon (Salmo salar). Aquaculture 433, 295–298. Yañez JM, Naswa S, López ME, Bassini L, Cabrejos ME, Gilbey J, Bernatchez L, Norris A, et al (2014) Development of a 200K SNP Array for Atlantic Salmon: Exploiting Across Continents Genetic Variation, in: 10th Wcgalp. Vancouver, Canadá. Yáñez JM, Barría A, Oyarzún M, Neira R, Newman S, Lhorente JP (2015) Negative genetic correlation between resistance against Piscirickettsia salmonis and body weight in coho salmon (Oncorhynchus kisutch). Aquaculture (Submitted).
















