El virus ISA fuera de la balsa jaula
González RR.1,2, Ruiz P.1, Llanos-Rivera A.1, Cruzat F.1,2, Silva J.1 1 Unidad de Biotecnología Marina, Facultad de Ciencias Naturales y Oceanográficas, Universidad de Concepción, Chile. Casilla 160-C, Concepción, Chile. 2 Centro de Investigación Oceanográfica del Pacífico Sur-Oriental (Fondap-Copas), Copas Sur Austral, Universidad de Concepción. Casilla 160-C, Concepción, Chile.Basado en el manuscrito publicado en Aquaculture 318 (2011) 37-42
Introducción La anemia infecciosa del salmón (ISA) fue reportada por primera vez en Noruega en el año 1984, en peces provenientes de hatchery de salmón Atlántico (Salmo salar Linnaeus, 1758) (Thorud & Djupvik, 1988). Posteriormente, la enfermedad ha sido reportada en Canadá, en el año 1996 (Mullins y col., 1998); en Escocia durante 1998 (Rodger y col., 1998); Chile, en 1999 (Kibenge y col., 2001a) y en USA, el 2000 (Bouchard y col., 2001). La información oficial disponible para nuestro país, según el Servicio Nacional de Pesca y Acuicultura (Sernapesca) y SalmonChile, indica que entre agosto del año 2007 y julio del 2009 se verificaron un total de 134 centros de cultivo en brote y 94 centros de cultivo sospechosos de la presencia de ISAv. Posteriormente, en el periodo 2010 a la fecha se han reportado otros 13 brotes (Sernapesca 2011).
Actualmente, en Noruega siguen existiendo brotes regulares de esta enfermedad. Según el “Norwegian Scientific Committee for Food Safety” los reservorios más probables de virus ISA son los propios salmones de cultivo (excretas) y algunos salmónidos silvestres, entre los que destacan la trucha café (Salmo trutta Linnaeus, 1758) y el propio Salmo salar. La importancia de estos reservorios en el origen de brotes de ISA indica que en el 30% de los casos corresponde a los centros de cultivo próximos, donde el virus puede ser arrastrado en la columna de agua adherido a material particulado o al microplancton. En el 70% de los brotes restantes, los reservorios podrían ser variados: (a) la infraestructura circulante en la industria salmonicultora (por ejemplo Wellboats); (b) peces silvestres que portarían variantes avirulentas que al contagiar a peces de cultivo se convierten a formas virulentas (reservorio sin cuantificación en Chile); y (c) transmisión vertical a lo largo del ciclo de vida provocando mortalidad en la fase marina (Norwegian Scientific Committee for Food Safety, 2007). El objetivo del presente trabajo fue determinar si existe la presencia del virus ISA en organismos silvestres que comparten el ambiente y fuentes de alimentación con los salmones cultivados en el ecosistema sur austral. Este estudio corresponde a la primera investigación en Chile que examina estos posibles reservorios, vectores y matrices ambientales por las que se podría diseminar y/o permanecer el virus ISA en este ecosistema.
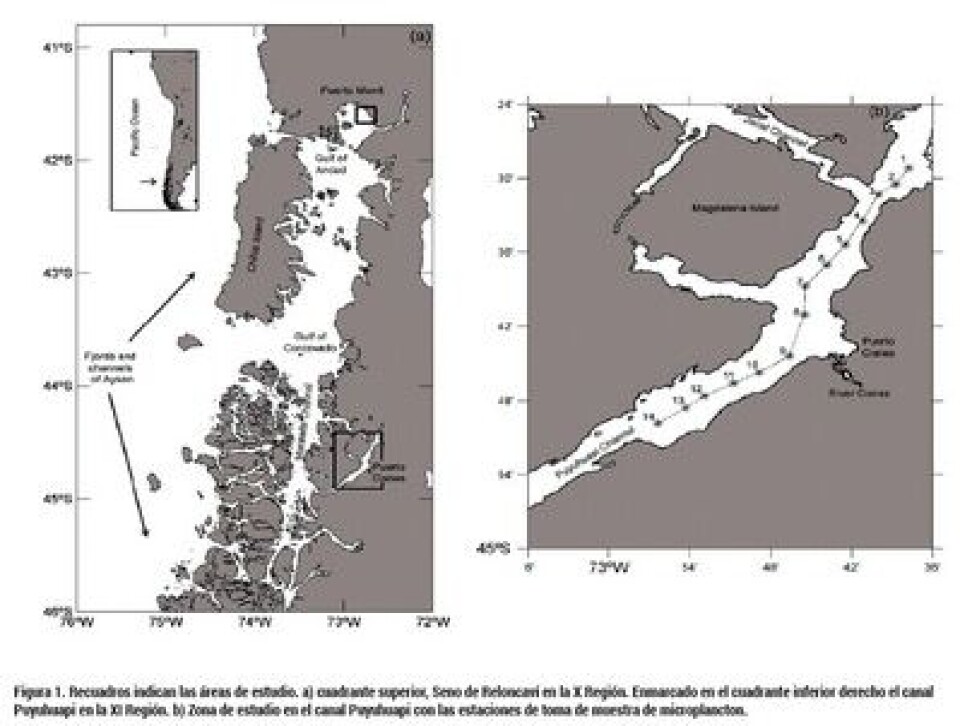
Materiales y métodos 1. Área de estudio Los organismos analizados provienen de la región de Los Lagos y la región de Aysén del General Carlos Ibáñez del Campo (Fig. 1). En la primer área (Seno de Reloncaví) se obtuvieron muestras de peces, moluscos y crustáceos. En la segunda área (Canal Puyuhuapi), se obtuvieron el mismo grupo de taxas, además muestras de microplancton y material fecal de lobo marino; Otaria flavescens (Shaw, 1800). 1.1 Obtención de muestras 1.1.1 Muestras de microplancton: las muestras fueron obtenidas desde la columna de agua del Canal Puyuhuapi (44º 19’ S, 72º 33’ O por el norte hasta 44º 57’S, 73º 21’O por el sur) en 14 estaciones de la columna de agua (Fig. 1). Las muestras de agua fueron obtenidas con botellas Niskin a 0, 10, 25 y 50 m de profundidad. Un litro de cada muestra fue tamizado por 100 μm, y posteriormente, filtrado a través de ester de celulosa de 47 mm de diámetro y 0,22 μm de poro en bajo vacío (< 100 mm de Hg). Este material fue mantenido congelado en nitrógeno líquido hasta su procesamiento y análisis en laboratorio. 1.1.2 Muestras de tejido: se analizaron 113 ejemplares, representando a 17 especies de distintas taxa. Para la mayoría de los ejemplares se contó con cuatro tejidos: corazón, músculo, riñón y branquia, conformando un total de 486 muestras. Las muestras obtenidas fueron almacenadas en RNAlater ® y congeladas a -80 °C hasta su posterior análisis. Las especies capturadas por localidad se encuentran contenidas en la Tabla 1. 2- Síntesis de cDNA y Transcripción Reversa- Reacción en cadena de la Polimerasa (RT- PCR): La mezcla de reacción para la transcripción reversa (RT) se realizó mezclando los siguientes componentes: 5 μL de buffer MMLV 5X, 10 mM final de cada dNTPs y 100 UI de la enzima transcriptasa reversa (Promega M-MLV Reverse Transcriptase) en un volumen final de 11 μL. Las muestras de cDNA obtenidas se sometieron a una amplificación por reacción en cadena de la polimerasa (PCR). A 2 μL de cDNA se le adicionó 20 pmoles de cada partidor forward y reverse para la amplificación específica de los segmentos 6 (Kibenge y col., 2007) y 8 del virus ISA (OIE 2006), buffer Taq 2,5X (Promega), MgCI2 2 mM, dNTPs 1mM (Promega), GoTaq Flexi DNA polimerasa (Promega) en presencia de DMSO 3%. 3- Amplificación del segmento 8 de ISAv por PCR en tiempo real y determinación de carga viral: se elaboró una curva de calibración con diluciones de un plásmido purificado que contenía el inserto del segmento 8 del virus. Las diluciones evaluadas estaban entre 1x10 hasta 1 copias/μL. La mezcla de amplificación contenía 10μL de TaqMan Universal PCR Master Mix 2X, 900 nM de cada partidor correspondiente al segmento 8 (Snow y col., 2006), 1,2 μL cDNA y 0,8 μL de sonda específica (Snow y col., 2006) en su concentración óptima. El programa de amplificación por RT-PCR en tiempo real utilizado fue: 40 ciclos a 95 ºC por 10 minutos, 95 ºC por 15 segundos y 60 ºC por 1 minuto. Para el análisis de los datos y determinación de la eficiencia de la curva de calibración se utilizó el programa Rotor Gene series Software 1.7. 4- Secuenciación y Análisis Bioinformático: la secuenciación de los plásmidos purificados que contenían el segmento 6 clonado se realizó por la empresa de servicios Macrogen (Korea). Las secuencias obtenidas se limpiaron y posteriormente fueron alineadas con secuencias existentes para el segmento 6 de ISAv publicadas en GenBank, utilizando el software gratuito de alineamiento múltiple, denominado ClustalW. 5- Calculo del factor de condición: el factor de condición fue calculado según Fulton (1902) de la siguiente manera: K=( P/L³) x 100 ; donde P, es el peso del pez, y L la longitud.
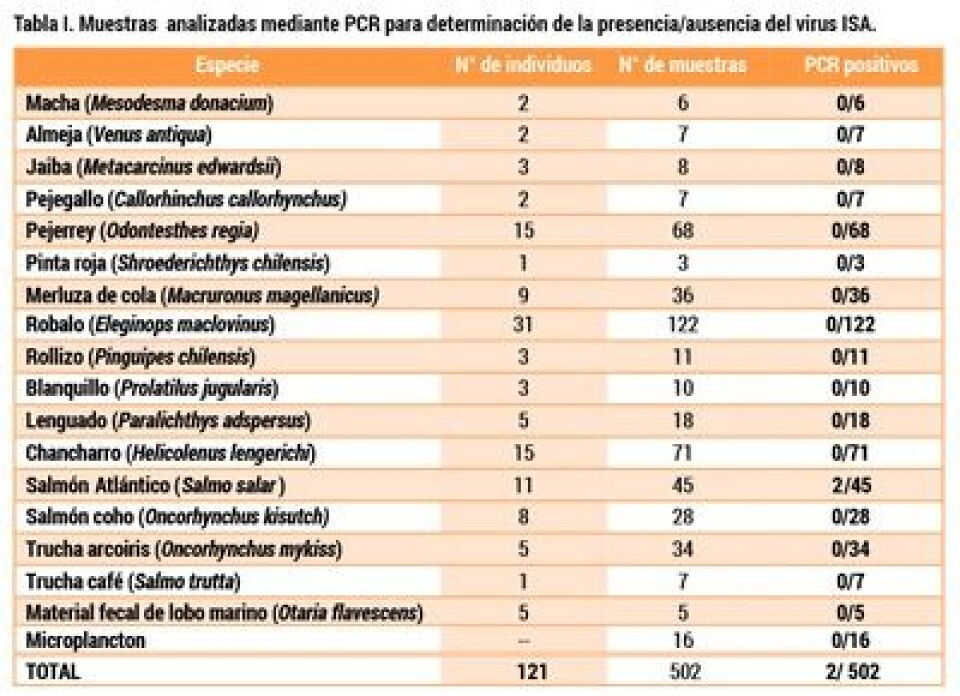
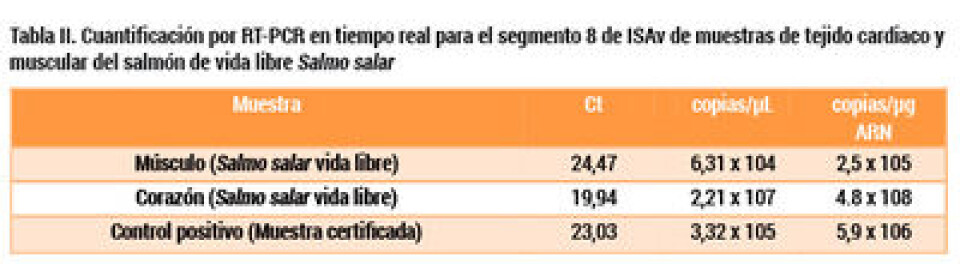
Resultados 1. Detección del segmento 6 y 8 del genoma del virus por RT-PCR convencional en peces, crustáceos, moluscos, fecas de lobo marino y microplancton. Con la excepción de los salmónidos, todas las muestras analizadas provenientes de los organismos capturados y matrices colectadas en las áreas de estudio, así como también las muestras de microplancton provenientes de la columna de agua resultaron ser negativas para la amplificación del segmento 6 y 8 del virus (Tabla 1). En las muestras obtenidas de salmónidos de vida libre se detectó la presencia del segmento 6 y 8 del virus ISA en Salmo salar. Un individuo de esta especie obtenido desde el Canal Puyuhuapi, fue positivo tanto en tejido cardiaco como en tejido muscular. La amplificación del segmento 6 del virus para estas muestras arrojo un fragmento de aproximadamente 1000 pb. La secuencia de este fragmento purificada a partir de los tejidos analizados se utilizó en el posterior análisis filogenético y la cuantificación del ARN viral. De los tejidos analizados, el corazón mostró una carga viral dos órdenes de magnitud mayor comparado con una muestra proveniente de un centro de cultivo en brote certificada y utilizada como control positivo (Tabla 2). Finalmente, el factor de condición para el salmón infectado calculado entrega un valor K = 1,23.
2. Análisis filogenético. El análisis filogenético de las secuencias nucleotídicas de los clones obtenidos a partir de los cDNA positivos al virus provenientes de las muestras del Salmo salar de vida libre infectado, fueron traducidas y comparadas con las secuencias de aminoácidos para la región HPR del segmento 6 de ISAv disponibles en la base de datos Genbank: cepa europea EU118820 de HPR0; Markussen y col., 2008) y cepa chilena AM941715 de HPR7b (Godoy y col., 2008). El alineamiento de aminoácidos de todas las muestras obtenidas revela un 100% de identidad con la variante genética europea HPR7b (Fig. 2).
Discusión Una de las características básicas de las infecciones virales es que son especie- específicas debido al reconocimiento molecular de las proteínas virales por los receptores de membrana de la célula hospedadora (Nelson & Cox, 2001). No obstante, se ha comprobado que el virus ISA, además de ser capaz de provocar la enfermedad principalmente en salmón Atlántico, también puede infectar a otras especies de salmónidos cultivados (Norwegian Scientific Committee for Food Safety, 2007). Fuera de la familia Salmonidae, en el hemisferio norte se ha detectado la presencia, pero sin sintomatología clínica del virus, en especies nativas (Stagg y col., 2001; Rolland, 2004) y en varias especies posterior a desafíos experimentales frente a la infección en condiciones de laboratorio (Raynard, 2001; Snow y col., 2001, Kibenge y col., 2001b; Nylund y col., 2002; Rolland y Winton, 2003; Plarre y col., 2005). Así también, información no publicada apunta a que ISAv puede mantener su actividad en agua de mar no estéril durante días o bien unida a superficies como cascos de embarcaciones y balsas jaulas (Norwegian Scientific Committee for Food Safety, 2007). En este sentido, existe una solicitud de patente al Instituto Nacional de Protección Industrial de Chile, para un “Método para la concentración de virus ISA desde la columna de agua” (Solicitud CL162- 2011), el que puede ser una herramienta clave en la evaluación de presencia de este virus en la columna de agua de áreas de producción acuícola como los barrios hoy existentes. Los resultados de este estudio, demuestran la ausencia del virus ISA en todos los individuos de las 13 especies nativas examinadas y la presencia del virus en una de las cuatro especies de salmónidos de vida libre capturados. Las secuencias aminoacídicas del gen de Haemaglutinina (segmento 6) obtenidas desde los tejidos infectados de Salmo salar de vida libre, alineadas con las secuencias de la región HPR0 y HPR7 (disponibles en Genbank), indican que este organismo era portador de la cepa perteneciente al grupo HPR7b, considerada la de más alta virulencia, basada en el mayor porcentaje de recurrencia de esta cepa encontrada en los brotes que han ocasionado mortalidades masivas (Kibenge y col., 2007; Kibenge y col., 2009). La cuantificación del ARN viral en el individuo infectado revela que este Salmo salar de vida libre tenía una carga viral dos órdenes de magnitud mayor que los salmónidos de cultivo infectados provenientes de una muestra certificada utilizada como control positivo en este trabajo (Tabla 2). En otras palabras, el ejemplar de Salmo salar capturado portador de la infección poseía en el momento del muestreo una alta carga viral de la cepa más virulenta determinada en Chile (HPR7b), no obstante, en el individuo no se observó sintomatología clínica aparente, más aun, el valor de su factor de condición (K = 1,23) indica que el espécimen se encontraba en buena condición y creciendo (Fulton, 1902; Nikolsky, 1963). Es importante considerar que en el hemisferio norte los salmónidos corresponden a fauna nativa y son considerados un vector activo en la propagación de la epidemia, en especial S. salar y S. trutta, por lo tanto, este género está siendo ciertamente considerado dentro de los supuestos epidemiológicos de la dispersión del virus ISA, así como también su relación con la fauna nativa (Norwegian Scientific Committee for Food Safety, 2007). En el caso de Chile, si bien en cierto que la presencia de salmónidos de vida libre puede ser asumida como producto de los organismos escapados provenientes de centros de cultivo, se debe considerar que existe evidencia a favor de que especies de Salmónidos, como el salmón chinook, podrían realizar su ciclo vital completo en este ambiente (Astorga y col., 2008). De esta manera, ambos orígenes podrían ser reservorios y vectores activos plausibles involucrados en la diseminación de la enfermedad en el ecosistema sur austral. En este estudio, también se analizaron otras matrices ambientales donde podría existir la presencia del virus, como material fecal de lobo marino (Otaria flavescens) y microplancton. Aunque ambas matrices resultaron ser negativas a la presencia del virus ISA, se ha confirmado que el virus puede mantenerse activo en distintos ambientes y condiciones fisicoquímicas (Norwegian Scientific Committee for Food Safety, 2007). También se ha señalado que la acidez del sistema digestivo neutraliza el efecto del virus impidiéndose con ello la infección, sin embargo, estudios experimentales en células SHK-1 donde se ha acidificado el medio durante la inoculación, demuestran un aumento en la cantidad de virus producido por las células (pH 4,0). Esto demostró que a pH bajo no sólo se aumenta la asociación y la fusión, sino también se aumenta la producción de virus infeccioso (Eliasssen y col., 2000). En este mismo sentido, además de los lobos marinos, sería interesante incorporar aves como potencial objeto de estudio, tanto carroñeras como pescadoras, ya que las presas de salmón son parte de su alimentación y no existen antecedentes en favor o en contra sobre su participación como vectores activos en los patrones de distribución involucrados en la diseminación de ISAv. Se debe destacar que el presente estudio, aunque abarca un número total de 108 ejemplares examinados, provenientes de 17 especies, incluidos salmónidos de vida libre, están lejos de los 50.000 ejemplares estudiados en el hemisferio norte (Rolland, 2004). Es por esta razón, que se hace necesario ampliar el número de individuos y especies que permitan descartar o confirmar más robustamente su presencia en la fauna nativa del ecosistema sur austral u organismos de vida libre, no nativos, dada la importancia que estos podrían tener en la dinámica espacio-temporal de la epizootia. Actualmente, en Chile se ha establecido un Plan de Contingencia para la Anemia Infecciosa del Salmón, basado en la experiencia internacional y los lineamientos de la Organización Mundial de Sanidad Animal (OIE), con una serie de medidas orientadas a la contención de los brotes, tales como el sacrificio de los organismos de las jaulas infectadas, delimitación de zonas de cuarentena y vigilancia, restricción de movimientos de la zona afectada, estrictas medidas de bioseguridad, condiciones de cosecha y proceso especiales para centros ubicados en las zonas bajo cuarentena y vigilancia, establecimiento de barrera sanitaria para las regiones XI y XII, y monitoreo y vigilancia permanente de los centros de las zonas afectadas y otros relacionados con el brote (Sernapesca, 2008). Sin embargo, ninguna de estas medidas contemplan en alguna de sus partes la interacción del virus con otros componentes activos del ecosistema, como por ejemplo, la fauna nativa o los salmónidos de vida libre, donde el presente trabajo es el primer reporte que demuestra su presencia a nivel molecular en el ecosistema sur austral de Chile.

Agradecimientos Este trabajo fue financiado por el Proyecto FIC-2008 (Gore, Aysén) “Determinación de ISAv en especies nativas y desarrollo de una línea celular para su propagación y experimentación” (Gore, Aysén) y por el Programa Copas Sur Austral (PFB-31/2007). Los autores desean expresar su agradecimiento al director ejecutivo del Ciep Dr. Giovanni Daneri y su grupo de trabajo, especialmente a Don Juan Ramón Velásquez. Así también, a la empresa Biovac por proveer los controles positivos, a Don Rodrigo Quiróz de la Municipalidad de Puerto Cisnes y a Don Roberto Velásquez por la colaboración a este trabajo.
Literatura citada Astorga M, C Valenzuela, I Arismendi & JL Iriarte. 2008. Naturalized Chinook salmon in the northern Chilean Patagonia: Do they originate from salmon farming?. Revista de Biología Marina y Oceanografía 43(3): 669-674. Bouchard D, G Brockway, C Keleher & P Merrill. 2001. First report of infectious salmon anaemia (ISA) in the United States. Bull Eur Assoc Fish Pathol 21: 86-88. Fulton T. 1902. Rate of growth of sea-fishes. Sci. Invest. Fish. Div. Scot. Rept.1: 22-20. Godoy M, F Kibenge, A Aedo, M Kibenge, D Groman, H Grothusen, A Lisperguer, M Calbucura, F Avendaño, M Imilán & M Jarpa. 2008. Primera detección, aislamiento y caracterización molecular de ISA-v en salmón del Atlántico (Salmo salar) de cultivo en Chile. SalmoCiencia 2: 47-55. Kibenge FS, MJ Kibenge, PK McKenna & P Stothard. 2001a. Antigenic variation among isolates of infectious salmon anaemia virus correlates with genetic variation of the viral haemagglutinin gene. J Gen Virol 82: 2869-2879. Kibenge F, O Garate, G Johnson, R Arriagada, M Kibenge & D Wadowska. 2001b. Isolation and identification of infectious salmon anaemia virus (ISAv) from Coho salmon in Chile. Dis. Aquat. Org. 45:9-18. Kibenge FS, H Xu, MJ Kibenge, B Qian & T Joseph. 2007. Characterization of gene expression on genomic segment 7 of infectious salmon anaemia virus. J Virol 4: 34. Kibenge F, M Godoy, Y Wang, M Kibenge, V Gherardelli, S Mansilla, A Lisperger, M Jarpa, G Latorre, F Avendaño, M Lara & A Gallardo. 2009. Infectious salmon anaemia virus (ISAv) isolated from the ISA disease outbreaks in Chile diverged from ISAv isolates from Norway around 1996 and was disseminated around 2005, based on surface glycoprotein gene sequences. Virology Journal 6: 88. Markussen T, CM Jonassen, S Numanovic, S Braaen, M Hjortaas, H Nilsen & S Mjaaland. 2008. Evolutionary mechanisms involved in the virulence of infectious salmon anaemia virus (ISAv), a piscine orthomyxovirus. Virology. 10;374(2): 515- 27. Monier A, J Claverie & H Ogata. 2008. Taxonomic distribution of large DNA viruses in the sea. Genome Biology 9: 106-118. Mullins J, D Groman & D Wadowska. 1998. Infectious salmon anaemia in salt water Atlantic salmon (Salmo salar) in New Brunswick, Canada. Bull Eur Assoc Fish Pathol 18: 110-114. Nelson D & Cox M. 2001. Principios de Bioquímica de Lehninger. En: Omega (ed) . pp 46-47. Barcelona, España. Nikolsky G.V. 1963. Biological basis of mathematical simulation of fish population dynamics. Vopr. Ikhtiol. 4: 1-29.3. Nylund A, M Devold, J Mullins & H Plarre. 2002. Herring (Clupea harengus): A host for infectious salmon anaemia virus (ISAv). Bull. Eur. Assoc. Fish Pathol. 22: 311-318. Norwegian Scientific Committee for Food Safety. 2007. Which risk factors relating to spread of Infectious Salmon Anaemia (ISA) require development of management strategies?. OIE (Organización Mundial de Sanidad Animal). 2006. Manual de Pruebas de Diagnóstico para los Animales Acuáticos, 5a edición, Paris. [en línea] <http://www.oie.int. > Plarre H, M Devold, M Snow & A Nylund. 2005. Prevalence of infectious salmon anaemia virus (ISAv) in wild salmonids in western Norway. Dis. Aquat. Org. 66: 71- 79. Raynard RS, AG Murray & A Gregory. 2001. Infectious salmon anaemia virus in wild fish from Scotland. Dis. Aquat. Org. 46: 93-100. Rodger H, T Turnbull, F Muir, S Millar & R Richards. 1998. Infectious salmon anaemia (ISA) in United Kingdom. Bull Eur Assoc Fish Pathol 18: 115-116. Rolland JB & JR Winton. 2003. Relative resistance of Pacific salmon to infectious salmon anaemia virus. J. Fish Dis. 26: 511-520. Rolland JB. 2004. Studies of factors affecting the epizootiology of infectious salmon anemia (ISA). Thesis Dr. Scient. University of Bremen. Sernapesca (Servicio Nacional de Pesca). 2008. Balance de la situación sanitaria de la anemia infecciosa del salmón en chile de julio del 2007 a julio del 2008. En: Sernapesca, Unidad de Agricultura, 31pp. Sernapesca (Servicio Nacional de pesca ). 2009. Anuario 2009. [en línea] <http://www.sernapesca.cl/index.php?option=com_remository&Itemid=54&func=fileinfo&id=4292 Snow M, RS Raynard & DW Bruno. 2001. Comparative susceptibility of Arctic char (Salvelinus alpinus), rainbow trout (Oncorhynchus mykiss) and brown trout (Salmo trutta) to the Scottish isolate of infectious salmon anaemia virus. Aquaculture 196:47- 54. Snow M, P McKay, AJA McBeath, J Black, F Doing, R Kerr, CO Cunningham, A Nylund & M Devold. 2006. Development, application and validation of a taqman real-time RT-PCR assay for the detection of infectious Salmon Anaemia Virus (ISAv) in atlantic salmon (Salmo salar). Vannier P. Espeseth D (eds); New Diagnostic Technology:applications in animal health and biologics controls. Dey Biol. (basel). Basel. Karger 126: 133-145. Stagg RM, DW Bruno, CO Cunningham, RS Raynard, PD Munro, AG Murray, CE Allan, DA Smail, AH McVicar & TS Hastings. 2001. Epizootiological investigations into an outbreak of infectious salmon anaemia (ISA) in Scotland. Report nº 13/01, FRS Marine Laboratory, Aberdeen. Thorud T & H Djupvik. 1988. Infectious salmon anaemia in Atlantic salmon (Salmo salar). Bull Eur Assoc Fish Pathol 8: 109-111.
















