La caligidosis, una parasitosis que complica a la salmonicultura chilena
Aprendiendo Acuicultura es una serie de artículos destinada a quienes tienen una participación activa en la industria acuícola. Esta sección está organizada con el propósito de entregarle información que esperamos encuentre útil en su trabajo diario, pues cuanto más conocimiento tenga, mejor podrá optimizar las condiciones de los peces y, un mejor bienestar animal, conduce a un desempeño más productivo y sustentable. Quisiéramos recibir sus observaciones y comentarios acerca de esta serie y apreciaremos sus sugerencias para futuros temas que le gustaría ver abordados aquí. Por favor envíenos sus comentarios a contacto@salmonxpert.cl
La sección Aprendiendo Acuicultura está escrita y editada por Gladys Asencio, Magister en Ciencias en Ecología Marina, especialista en ecología de microcrustáceos e investigadora del Centro de Investigaciones I-Mar de la Universidad de Los Lagos. Puede contactarla en la siguiente dirección de correo electrónico: gasencio@ulagos.cl
III. Caracterización genética del parásito Este capítulo entrega una revisión de los estudios desarrollados en los últimos 20 años para caracterizar genéticamente las especies de calígidos que impactan sanitaria y económicamente a la salmonicultura, en Chile (Caligus rogercresseyi) y el hemisferio norte (Lepeophtheirus salmonis); así como los esfuerzos tecnológicos realizados para lograr a) la caracterización genética de las poblaciones presentes en diferentes zonas geográficas, buscando sus marcadores poblaciones, b) entender la relación parásito-hospedero a nivel molecular, y c) obtener una vacuna para su control. Además se presentan los avances de las investigaciones en caligidosis que están desarrollando tres grupos de genetistas Chilenos.
1.- Lepeophtheirus salmonis Estudios sobre genética de L. salmonis realizados con diferentes métodos han mostrado que existe diferenciación en las poblaciones de esta especie, tanto en Noruega (Isdal y col. 1997) como en Escocia (Todd y col. 1997). Por lo tanto, existe una plasticidad fenotípica donde las enzimas se expresan de forma diferente durante las diferentes etapas de vida del parásito o como consecuencia de factores ambientales (Isdal y col., 1997). Datos de microsatélites de seis loci mostraron variaciones significativas entre las poblaciones de L. salmonis del océano Pacífico y de océano Atlántico (Todd y col. 2004). Del mismo modo, Tjensvoll y col. (2005) reportaron diferencias en el genoma mitocondrial entre una población de L. salmonis de Japón y de océano Atlántico. Posteriormente, a partir de muestras obtenidas de nueve poblaciones en todo el océano Pacífico, se encontró que los genes nucleares difieren en promedio en un 3,2% y el genoma mitocondrial en un 7,1% (Yazawa y col. 2008). Estos hallazgos son consistentes con el aislamiento geográfico y la divergencia ocurrida en salmones pertenecientes a los géneros Oncorhynchus y Salmo hace 11-24 millones de años (McKay y col. 1996). Trabajos realizados por Tjensvoll y col. (2006) sobre poblaciones de L. salmonis en salmón chum silvestre (Oncorhynchus keta) del océano Pacífico (Ishikari, Japón) demostraron que estas poblaciones eran distintas a las de Noruega, Escocia, Rusia y Canadá. Analizando cuatro genes mitocondriales (A6, COI, Cyt B y 16S rRNA). Esto sugiere un aislamiento geográfico o una total reducción del flujo de genes entre L. salmonis del océano Pacífico y el Atlántico Norte. Esto podría ser explicado por una posible adaptación de esta especie al hospedador Oncorhynchus, debido a que en el Atlántico Norte parasitan a los géneros Salmo y Salvelinus, mientras que las especies presentes en el océano Pacífico se encuentran en su mayoría dentro de los géneros Oncorhynchus (Nagasawa y Takami 1993, Nagasawa 2004). La secuenciación del genoma de piojos de mar, y de copépodos en general, puede ser utilizada para estudiar genes importantes para el desarrollo de éstos. Con respecto a este tipo de estudios, aparece actualmente la tecnología de ARN de interferencia, que se ha utilizado con éxito en L. salmonis (Dalvin y col., 2009; Campbell y col., 2009). Otros estudios están dirigidos al componente genético en la resistencia a los piojos de mar en el salmón del Atlántico. Por ejemplo, sobre los efectos de sustitución de alelos que sugieren que existen SNPs asociados con la resistencia genética. Gharbi y col. (2009) ya han detectado QTL (locus de un carácter cuantitativo) para la resistencia a los piojos del salmón. Los investigadores utilizaron tres familias de salmones que mostraron tener asociaciones significativas entre los recuentos de piojos y la variación de marcador en la UBA, DAA o ambos (Glover y col. 2007).
2.- Caligus rogercresseyi
La caracterización genética de C. rogercresseyi se encuentra en desarrollo, registrándose a partir del 2003 varias secuencias del gen ribosomal 18s en la base GenBank (Andree y Zagmutt, 2003), correspondientes a parásitos colectados en truchas de cultivo en la zona de Maillen en la X región. Posteriormente, se han desarrollado varias investigaciones para analizar: 1) la genómica de la especie y otros calígidos de nuestras costas, 2) la variabilidad poblacional del parásito en todas las zonas en donde se encuentra presente, tanto en la X como XI región, 3) la expresión génica en distintas condiciones, como exposición a un patógeno, tratamientos antiparasitarios, nutrición y las distintas etapas del ciclo de vida de esta especie, 4) la opción de formular una vacuna para su control. A continuación, se desarrollarán cada una de estas temáticas, mediante resúmenes que presentan los investigadores encargados de cada una de ellas.
2.1.- Plataforma genómica de la diversidad de Calígidos presente en peces de la costa chilena. M. Teresa González1,2; Zambra López1; Raúl Castro3. 1Laboratorio de Ecología y Epidemiología Marina (LEPyEM); 2Instituto Ciencias Naturales, Alexander von Humboldt; 3Depto. Recursos Naturales y Cs. Ambientales Facultad de Ciencias del Mar y Recursos Biológicos, Universidad de Antofagasta. Angamos 601-Antofagasta. Email: teresa.gonzalez@uantof.cl Los parásitos pueden poner en peligro la sustentabilidad, éxito y expansión de un cultivo de cualquier especie. Por lo tanto, es crucial identificar correctamente las especies parásitas locales y determinar cuáles son sus potenciales efectos dañinos. Los copépodos calígidos (Caligus spp. y Lepeohtheirus spp.) o vulgarmente conocidos como piojos de mar (Sea lice, en inglés) infestan un amplio rango de peces y tienen la capacidad de cambiar sus tasas de transmisión con la incorporación de un nuevo hospedador en el sistema, como ha ocurrido con C. rogercresseyi en el sur de Chile (González y col. 2012) y con L. salmonis en el hemisferio norte (Costello 2006). En peces de la costa chilena se reconocen ocho especies de Caligus (C. cheilodactylus, C. teres, C. rogercresseyi, C. aesopus, C. bonito, C. debueni, C. lalandei, C. quadratus) y siete especies de Lepeophtheirus*(L. chilensis; L. frecuens; L. mugiloides; L. zbigniewi, L. dissimulatus; L. yañezi; L. edwarsi) (Muñoz y Olmos, 2007).
Entre las especies más generalistas se encuentran C. cheilodactylus infestando al menos ocho especies de peces a lo largo de la costa chilena y C. quadratus que infesta al menos cuatro especies de peces litorales en el norte. A la fecha, sin embargo, no se había realizado un estudio usando marcadores moleculares que permitan corroborar la identidad de las especies de Caligus y Lepeophtheirus infectando a las distintas especies de peces litorales de nuestra costa. En nuestro laboratorio, con financiamiento de distintos proyectos (ver abajo), hemos evaluado mediante secuenciación de los genes 18 S, región D1-D2 del gen nuclear 28 S y el gen mitocondrial Citocromo Oxidasa subunidad I (COI), la diversidad de especies de calígidos presente en especies de peces litorales, contrastando la morfología con secuencias genéticas.
Metodologías de extracción de DNA y PCR Para los individuos más pequeños (<2mm) la extracción de ADN se realiza agregando a cada tubo 500 μL de Chelex al 5% y 2,5 μL de Proteinasa K (20 mg/mL) (0,1 mg/mL proteinasa K). Luego se incuba a 60 °C durante cuatro horas y se lleva a ebullición por ocho minutos (Leung y col., 2009). Mientras que para los individuos de mayor tamaño, la extracción de ADN se realiza con un Kit de extracción E.Z.N.A.® para tejidos . Cada PCR obtiene un volumen final de 35 μL, utilizando 0,025 U/µL de Taq, 1X de Buffer, 0,2mM de dNTPs, 0,4P/µL de cada partidor (Forward y Reverse), 4mM MgCl2, 3,5 µL de DNA concentrado y se completa el volumen final con H2O. Se utilizan 2 PCR para amplificar el COI, en primera instancia, para el PCR-1, se utilizan los partidores de Folmer y col., 1994 (LCO1490 y HCO2198), cuyos parámetros del ciclo utilizado en el termociclador incluye una desnaturalización inicial de 95 °C (5 min), siguiendo con 40 ciclos de 95 °C (45 s), 50 °C (45 s) y 72 °C (1 min), para terminar con una fase de extensión final de 72 °C (10 min). Las muestras que resultan negativas con el PCR-1, se repiten con un PCR-2, cuyo partidores son obtenidos de Oines & Heuch (2005) (WOBCOIF y WOBCOIR) con un ciclo de desnaturalización inicial de 95 °C (2 min), siguiendo con 35 ciclos de 95 °C (30 s), 45 °C (30 s) y 72 °C (1 min) y, por último, una extensión final de 72 °C (2 min). El producto PCR se visualiza en un gel de agarosa de 1,5%. Posteriormente, se purifica usando el kit comercial E.Z.N.A.® Cycle Pure y se secuencia automáticamente en un secuenciador de electroforesis de capilar ABI 3730XL en la compañía Macrogen Inc., Corea del Sur (www.macrogen.com).
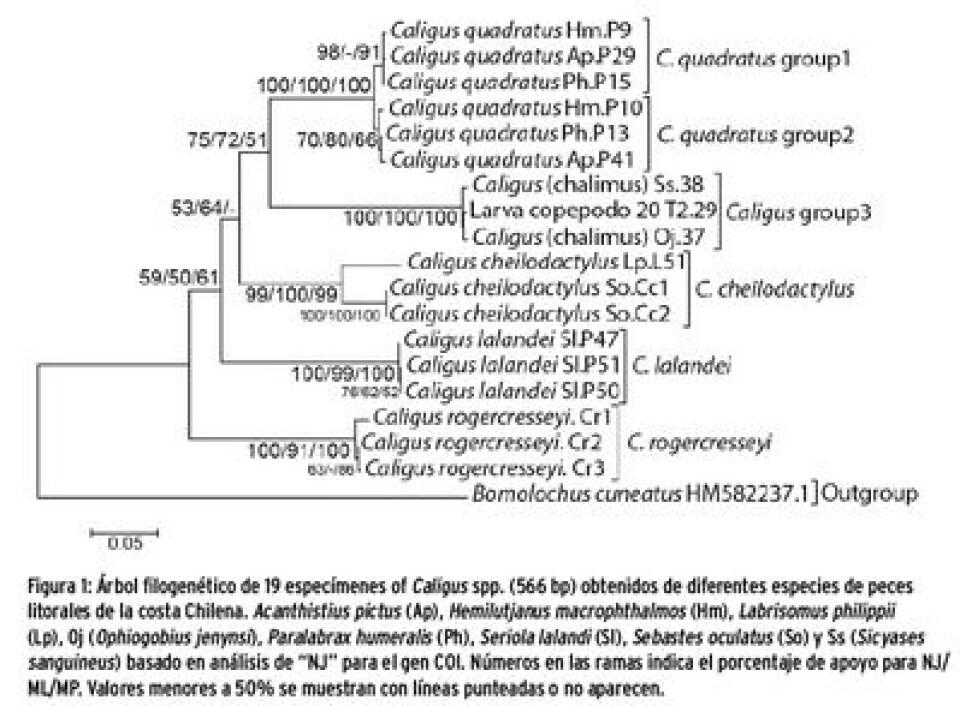
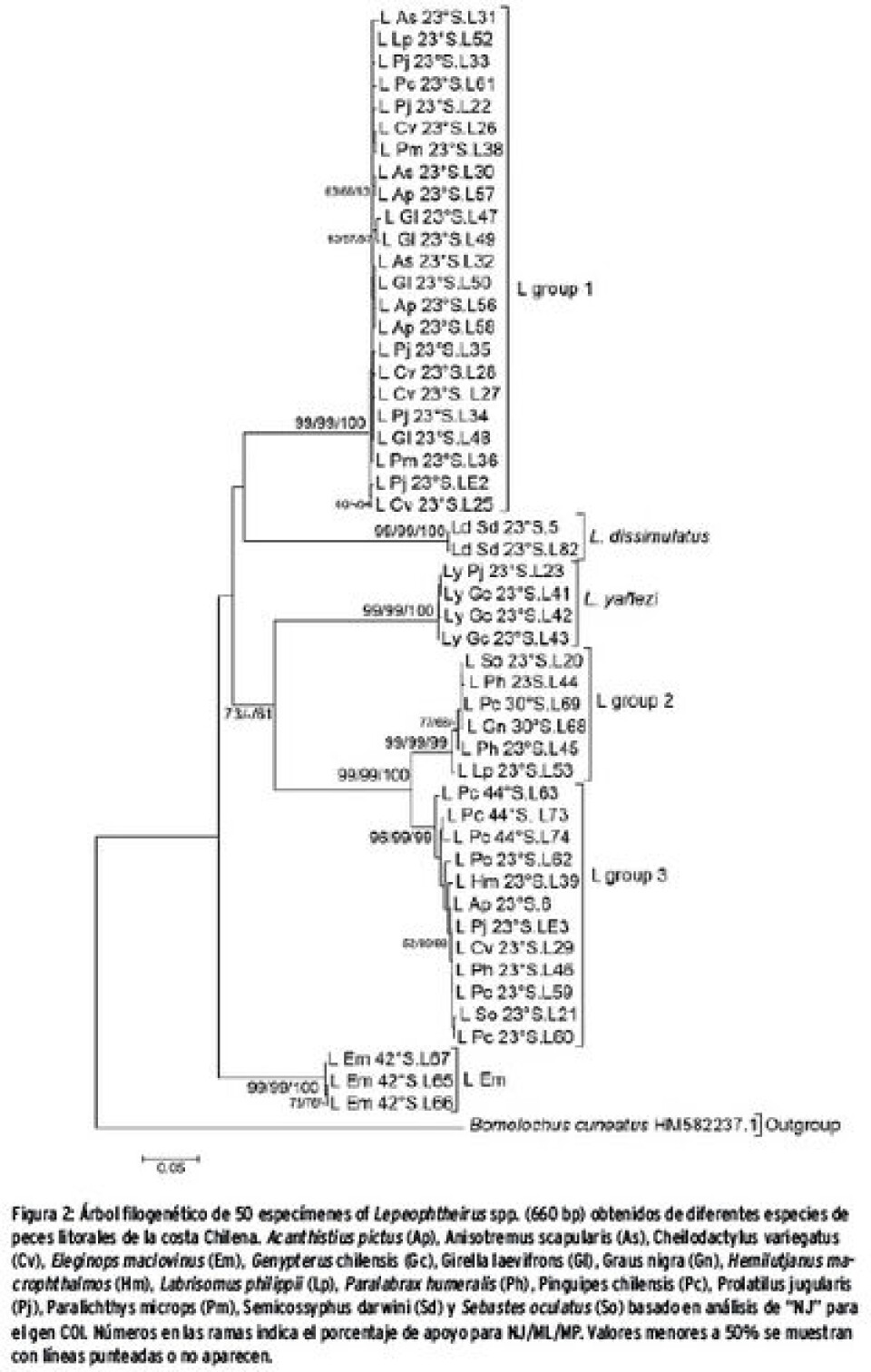
Análisis Moleculares: Las secuencias obtenidas se editan usando ProSeq v 3.0 (Filatov 2002) y se alinean con el programa Clustal 2 (Larkin y col. 2007). Los análisis para cada grupo (Caligus y Lepeophtheirus) se realizan en el programa Mega v.6 (Tamura y col. 2013), donde se utilizan los algoritmos de Neighbor-Joining (NJ) y Maximum Likelihood (ML), mientras que Maximum Parsimony (MP) se realiza en el programa PAUP* 4.0b10 (Swofford, 2003). Como grupo externo se utilizó Bomolochus cuneatus (HM582237.1) (Morales-Serna y col., 2013) obtenido desde la base de datos Genbank.

Resultados Caligus calloensis corresponde a un nuevo registro en peces sciaenidos de la costa norte de nuestro país. De las ocho especies de Caligus conocidas, la más común y que infesta a mayor número de especies de peces es C. quadratus, cuyos análisis moleculares muestran que corresponden a dos grupos de genotipos que co-ocurren en al menos ocho especies de peces con una distancia genética de 1,8%, menor que las comparaciones con las otras especies de Caligus (entre 22% y 34,9%). C. rogercresseyi no está presente en peces del norte de Chile y la hemos encontrado sólo en salmones cultivados y robalos del sur (Fig. 1).
De las 7 especies de Lepeophtheirus conocidas (Fig. 2), hay tres que son específicas: L. yañezi que infesta especies de Genypterus spp. (congrio negro y colorado), L. edwardsii que la hemos registrado sólo en Paralichtys spp. (lenguados) y L. dissimulatus registrada en Semmycosiphus maculatus (pejeperro). L. zbigniwei la hemos encontrado infestando peces, de hábitat intermareal y submareal en el centro y norte de Chile, pertenecientes a la familia Labrisomidae tales como Labrisomus phillipi (tomoyos) y Auchenionhus spp. Por otro lado, las especies L. chilensis, L. frecuens y L. mugiloides las hemos encontrado co-ocurriendo en diversas especies de peces litorales a lo largo de toda las costa chilena (20 °S a 45 °S). Las morfologías de L. chilensis y L. mugiloides son verdaderamente muy difíciles de distinguir, lo que también es concordante con la menor distancia genética entre estas dos especies (6%).
A la fecha, se reconocía sólo a L. mugiloides infestando al robalo, Eleginops maclovinus, pero nuestros resultados muestran que en este hospedador co-ocurre una segunda especie y se propone una nueva especie de Lepeophtheirus para la ciencia.
La plataforma genética que hemos ido generando en nuestro laboratorio LEPyEM nos permite identificar las diferentes especies de calígidos en distintos estados de desarrollo, y para aquellos estados adultos de especies que no presentan caracteres diagnósticos. Actualmente, en el marco de un proyecto Fondecyt 1120868, hemos registrado estadios larvales de copépodos infestando larvas de peces, cuyo escaso desarrollo de caracteres diagnósticos impide su identificación; y por lo tanto, estamos utilizando nuestras bases de datos con secuencias del gen Co-I, 18S ó 28S para contrastar con las secuencias de nuevas muestras y asignar así a las especies ya conocidas o reconocer especies nuevas para la ciencia.
Los proyectos que permitieron desarrollar estas investigaciones son: -CODEI (Convenio de Desempeño Universidad de Antofagasta, Código 5384): “Estudio de la diversidad de copépodos calígidos en peces costeros de la Segunda región y su potencial como agentes epidemiológicos: Desde la aproximación molecular hasta la dinámica poblacional. Investigador responsable: MT González -INNOVA-CORFO 09CNN14-5829: Determinación de las potenciales enfermedades parasitarias en acuicultura del dorado (Seriola lalandi) y otros peces en las costas de la Región de Antofagasta: Bases biológicas para el control y manejo de enfermedades. Investigador responsable: MT Gonzalez -FONDECYT 1120868. Parasite-fish interactions in their larval stages: What benefits are achieved by the parasites in too young hosts?. IR: Gabriela Muñoz; Co-Investigadores: M. Landaeta; MT González.
2.2.- Variabilidad del gen ribosomal 18S Rodrigo González 1, 2, Fernando Cruzat 1, 2, Gladys Asencio 3, Johanna Himelreichs 1, Pablo Oyarzún 1 y P. Lincoñir 1.

1 Unidad de Biotecnología Marina, Facultad de Ciencias Naturales y Oceanográficas, Universidad de Concepción, Casilla 160-C, Concepción, Chile; 2 Centro de Investigación Oceanográfica en el Pacífico Sur-Oriental (FONDAP-COPAS), COPAS Sur Austral, Universidad de Concepción, Casilla 160-C, Concepción, Chile; 3 Centro de investigación y desarrollo de recursos de ambientes costeros (Centro I-mar), Universidad de Los Lagos, Casilla 557, Puerto Montt, Chile. rogonzal@udec.cl
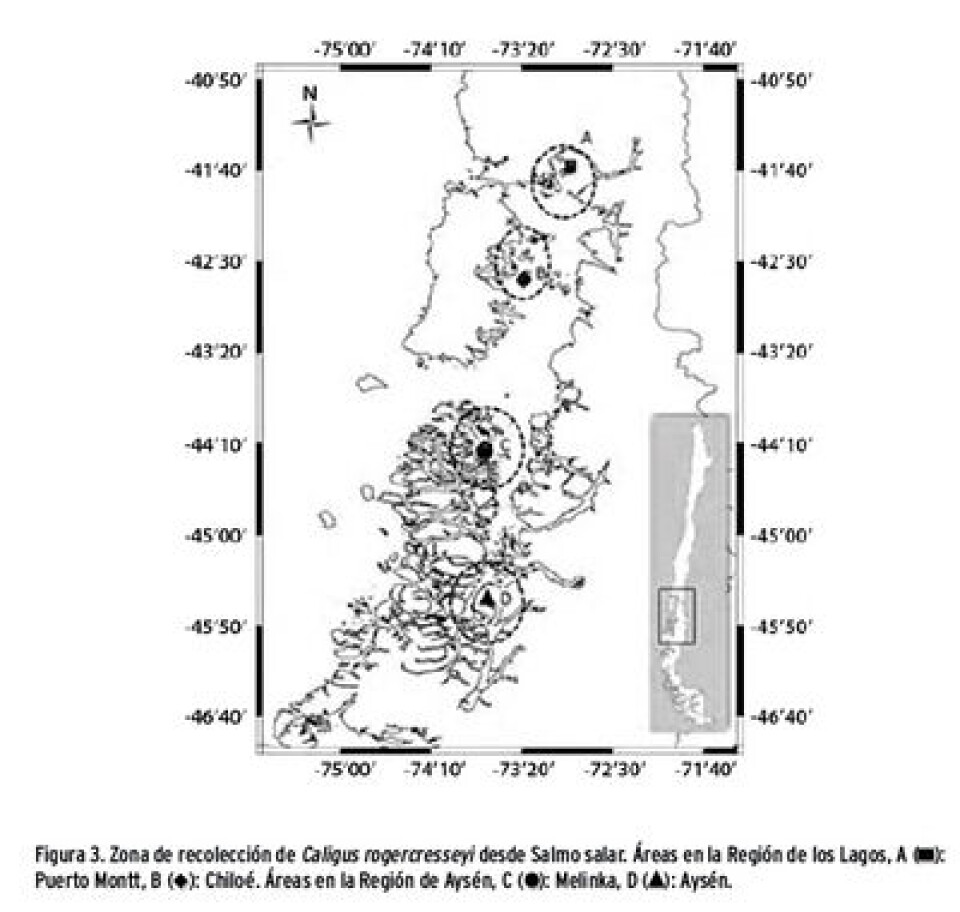
El objetivo del estudio fue caracterizar genéticamente el gen ribosomal 18S de C. rogercresseyi en peces de cultivo y determinar su variabilidad en este ectoparásito presente en la zona Sur-Austral de Chile. Para esto, se analizaron muestras correspondientes a C. rogercresseyi obtenidas durante los años 2012 y 2013 desde peces de cultivo (Salmo salar), en distintos centros de cultivo emplazados en cinco áreas del ecosistema Sur-Austral de Chile entre los 41 ºS y 45 ºS (Figura 3). La amplificación del gen ribosomal 18S en C. rogercresseyi se llevó a cabo mediante ensayo PCR convencional y los fragmentos amplificados se analizaron mediante electroforesis en gel de agarosa al 1,5%. La visualización de bandas se realizó por tinción con Bromuro de Etidio (0,5 µg/mL) y exposición en transiluminador UV. La purificación del fragmento de ADN del tamaño esperado (~ 700pb), se realizó mediante el kit comercial E.Z.N.A.® Gel extraction kit Omega Bio-Tek, según las recomendaciones del fabricante. El ADN purificado fue ligado al vector plasmidial pGEM®T-Easy Promega, utilizando una relación vector:inserto de 3:1 para favorecer la ligación, siguiendo las recomendaciones del fabricante. La transformación bacteriana se llevó a cabo utilizando células competentes Escherichia coli JM109® Promega y el producto de ligación, siguiendo la metodología de shock térmico. Las bacterias transformadas se sembraron en placas de agar con medio Luria-Bertani (LB). La confirmación de clones positivos se realizó mediante PCR. Las muestras de ADN plasmidial purificadas fueron secuenciadas por la empresa de servicios MACROGEN (Corea). Obtenidas las secuencias parciales del gen ribosomal 18S de C. rogercresseyi se realizó un análisis en la base de datos de NCBI usando el programa BLAST (http://www.ncbi.nlm.nih.gov/BLAST/) para verificar que los clones secuenciados corresponden a la especie C. rogercresseyi. Posteriormente, el análisis y la interpretación de las secuencias se llevó a cabo en el programa Vector NTI Advance® versión 10.3.0 InvitrogenTM y con el programa MEGA versión 5.05 (Tamura y col., 2011). Las secuencias obtenidas en centros situados en las distintas áreas geográficas seleccionadas se compararon con las bases de información registrados en el GenBank, estas últimas se detallan en la Tabla 1.
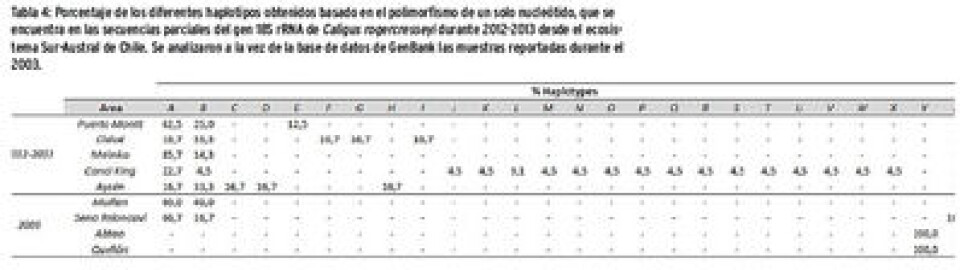
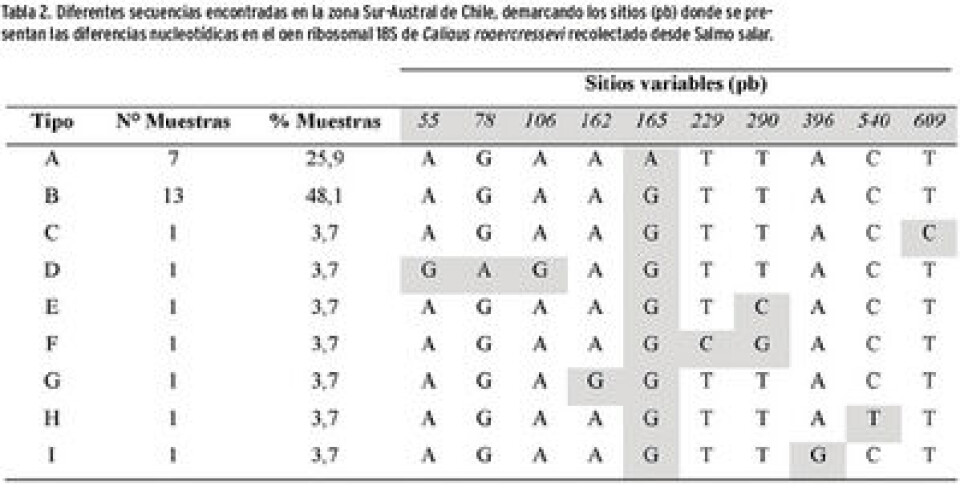
El alineamiento de las 27 secuencias analizadas en todas las áreas geográficas estudiadas (Tabla 2) mostró un 98,5% de posiciones similares, observándose nueve tipos de secuencias que presentaron variaciones nucleotídicas a lo largo del fragmento generado con los partidores diseñados para el gen ribosomal 18S. Los cambios variaron en porcentaje según la zona geográfica, como se muestra en la Tabla 3, destacando los centros de Aysén y Chiloé con la mayor variabilidad nucleotídica. El árbol filogenético construido con esta información y las secuencias del GenBank (Fig. 4) muestran que las secuencias del 2003 presentaron un 35% de similitud con las registradas en este estudio desarrollado entre el 2012-2013, permaneciendo así el 65% restante como nuevas. Posterior a este resultado, un año después se analizó nuevo material recolectado en diferentes centros de la región de Los Lagos y Aysén, registrándose una variación genética mayor a la reportada previamente, obteniendo 19 posiciones nucleotídicas variables entre las muestras secuenciadas, dando lugar a 17 haplotipos distintos (Tabla 4). Estas variaciones nucleotídicas fueron principalmente mutaciones de carácter transicional (Adenina por Guanina). Además, se pudo inferir que los cambios nucleotídicos han aumentado en el transcurso de un año, en comparación con los cambios ocurridos en años anteriores. Por ejemplo, Himelreich (2012) obtuvo una variación nucleotídica de 0,7% y que esta variación aumentó un 0,8% en diez años, mientras que en este trabajo se obtiene una variación de 2,6% de nucleótidos variables entre los haplotipos identificados, aumentando en un año a un 1,9%. Himelreich menciona la posición nucleotídicas 165 como la más significativa en cuanto a variaciones, concordando con este trabajo en el cual está posición varia en dos secuencias (B y O), siendo la posición más variable.
En este trabajo, sólo los haplotipos A y B, los cuales también fueron predominantes en el trabajo de Himelreich (2012), fueron previamente descritos, los demás haplotipos no habían sido reportados. Los haplotipos identificados en este trabajo no corresponden a haplotipos reportados antes de Himelreich (2012), esto se puede dar porque son nuevos haplotipos, o por falta de información genética en las bases de datos, por los escasos estudios llevados a cabo en materia de biología molecular aplicada a estos parásitos.
Este trabajo aumenta en un 34% las secuencias encontradas en la base de datos GenBank. Siendo que estas secuencias sólo se obtuvieron en un área geográfica limitada, como lo es Canal King. Esta gran variedad genotípica podría darse por importación de C. rogercresseyi desde otros sectores o por presión selectiva ambiental (incluida la utilización de fármacos antiparasitarios).
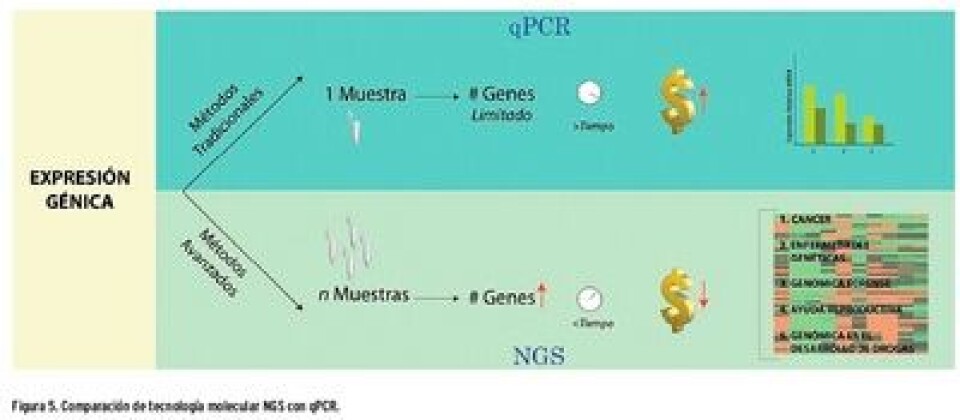
Cristian Gallardo-Escárate, Valentina Valenzuela, Gustavo Núñez, Jacqueline Chávez, Waleska Maldonado, Ana Teresa Gonçalves, Rodolfo Farlora, Diego Valenzuela. Laboratorio de Biotecnología y Genómica Acuícola, Centro Interdisciplinario de Investigación en Acuicultura – INCAR, Universidad de Concepción, Chile. E-mail: crisgallardo@udec.cl La búsqueda de marcadores biológicos para la comprensión de procesos biológicos complejos en especies modelos y no modelos, está actualmente experimentando avances sin precedentes. La inversión mundial en el desarrollo de tecnologías de alto rendimiento como secuenciación de última generación o “next generation secuencing (NGS)” ha revolucionado la investigación en plantas y animales permitiendo la secuenciación de genomas completos en sólo meses de trabajo de laboratorio. La tecnología NGS permite una exploración sistemática y comprensiva de las bases moleculares de distintos procesos biológicos, a través de la detección de genes funcionales y biomarcadores a un nivel masivo. De esta forma, actualmente existe la capacidad de generar una gran cantidad de datos, que en su conjunto representan una fracción significativa del genoma. NGS es una herramienta para la investigación genómica de punta y de alto impacto, ya que los costos de su aplicación disminuyen en forma constante y posee ventajas significativas con métodos tradicionales de evaluación de expresión génica como qPCR (Fig. 5). Esta tecnología ha sido aplicada en diferentes áreas de investigación, como por ejemplo; secuenciación de genotecas, identificación de marcadores moleculares como SNPs, evaluación genómica de drogas, perfiles de expresión de microRNA, así como detección temprana de enfermedades tanto en seres humanos como en especies de producción animal. Sin embargo, la aplicación más común de NGS es la generación de transcriptomas específicos, los cuales proporcionan acceso directo a las secuencias codificantes de miles de genes en forma simultánea y entregan información sobre sus niveles de expresión relativa. Dicha aproximación denominada RNA-seq permite determinar perfiles de expresión génica en distintas condiciones como exposición a un patógeno, nutrición o bien en distintas etapas del ciclos de vida de una especie. De este modo, RNA-seq permite la identificación de nuevos transcriptos, así como mutaciones puntuales con consecuencias funcionales específicas.
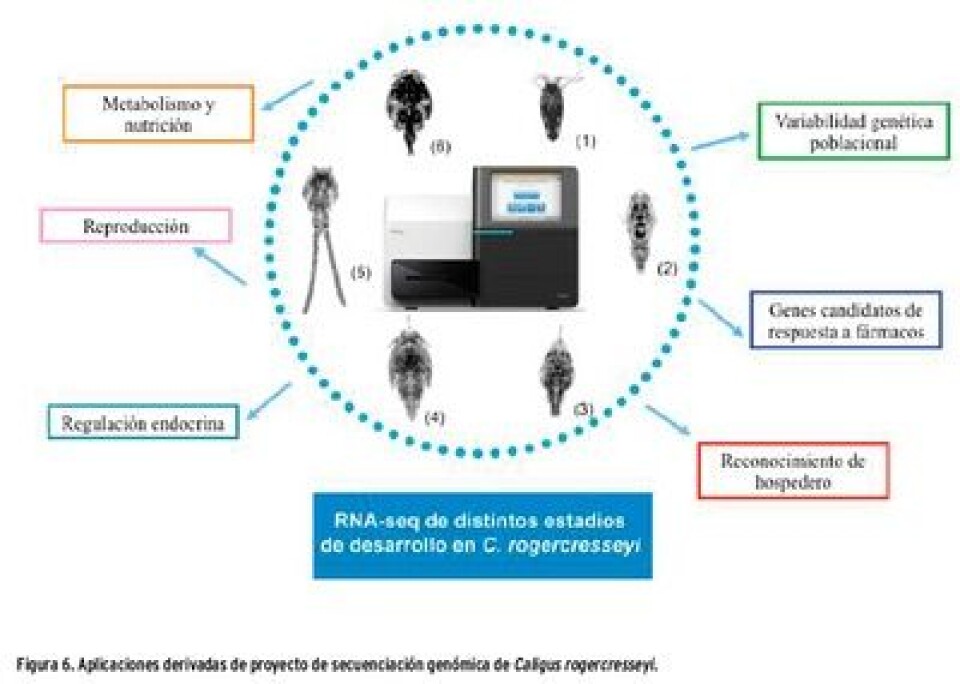
De esta forma, los avances tecnológicos en genómica permiten desarrollar una mirada integradora frente a distintos procesos biológicos complejos como crecimiento, reproducción, nutrición, metabolismo, respuesta inmunológica, estrés, entre otros. En este contexto, la iniciativa CALIGUS-seq desarrollada por Centro Interdisciplinario de Investigación en Acuicultura – INCAR, ha generado a través de un ambicioso proyecto transcriptómico más de 1.400 millones de secuencias transcritas desde ocho estadios de desarrollo del ciclo de vida de C. rogercresseyi (Nauplius I-II, copepodito, chalimus I-IV, hembras y machos adultos) [1] (Fig. 6).
A partir de esta información se han identificado sobre 84.000 genes. De ellos, 29.000 genes han sido relacionados a procesos biológicos claves (Fig. 6) como resistencia a fármacos (2-3), desarrollo larval y de sistema nervioso (4), respuesta inmune (5-6), metabolismo (7) y reproducción (8). Sin embargo, cerca de 55.000 genes no evidencian similitud con proteínas descritas previamente en base de datos públicas. Adicionalmente, se han caracterizados 71.116 polimorfismos de único nucleótido (SNPs) contenidos en 22.675 transcritos. Dicha información es relevante a fin de buscar relaciones entre dinámica poblacional y patogenicidad de C. rogercresseyi sobre salmones de cultivos. Por otra parte, los SNPs pueden ser utilizados para evidenciar genes específicos responsables de resistencia/susceptibilidad a fármacos utilizados en la industria salmonera como antiparasitarios [9]. Asimismo, la información generada permite la identificación de las bases moleculares involucradas en el reconocimiento de los peces hospederos [10]. El estudio genómico en C. rogercresseyi representa un hito relevante en investigación científica de excelencia, así como su aplicación en el diseño e implementación de nuevas estrategias de control de la caligidosis en Chile (Gallardo y col. 2013a, Valenzuela y Gallardo 2013a , Chávez y Gallardo 2013, Gallardo y col. 2013b, Valenzuela y Gallardo 2013b, Maldonado y Gallardo 2013, Gonçalves y col. 2013, Farlora y col. 2014, Núñez y Gallardo 2013, Núñez y col. 2014).
RESUMEN En siete capítulos se presentará una visión actualizada del conocimiento de los calígidos que impactan la industria salmonicultora mundial, con el objetivo de proporcionar herramientas para el mejoramiento de la vigilancia y manejo de esta enfermedad. En este artículo se presentó la caracterización genética de los calígidos de las costas chilenas, y una revisión de los avances en genómica de las dos especies de mayor impacto sanitario y económico para la salmonicultura mundial. La información documentada hasta hoy permite mostrar la variabilidad genética de ambas especies, así como de sus poblaciones en las áreas geográficas que habitan. Además, gracias al desarrollo tecnológico ocurrido en la última década, existen las herramientas de análisis que permitirán en el corto plazo generar la información necesaria para llevar a un mejor manejo de esta parasitosis en esta industria acuícola. En los próximos capítulos se analizarán las estrategias utilizadas para su vigilancia y control, así como nuevas alternativas que permitan un manejo sustentable de la industria salmonicultora mundial.
LITERATURA CITADA Andree, K.B. and Zagmutt, F. 2003. Genetic differences among populations of sea lice from farmed salmonids in southern Chile. , University of California, 1 Shields Ave., Davis, CA 95616, USA Boxaspen, K. 2006. A review of the biology and genetics of sea lice. e ICES Journal of Marine Science, 63: 1304-1316. Campbell, EM., C. Perl, AS Bowman. 2009. RNA-interference methods for gene-knockdown in the sea louse, Lepeophtheirus salmonis; studies on a putative prostaglandin E synthase. Parasitology 136: 867-874. Chávez, J y Gallardo C. 2013. Deltamethrin (AlphaMax) reveals modulation of genes related to oxidative stress in the ectoparasite Caligus rogercresseyi: implications on delousing drugs effectiveness. Aquaculture. Doi: 10.1016/j.aquaculture.2014.06.017. Costello, MJ. 2006. Review. Ecology of sea lice parasitic on farmed and wild fish. Trends Parasitol 22: 475-83. Dalvin, S., Frost P, Biering E, Hamre LA, Eichner C, Krossøy B, Nilsen F. 2009. Functional characterization of the maternal yolk-associated protein (LsYAP) utilizing systemic RNA interference in the salmon louse (Lepeophtheirus salmonis) (Crustacea: Copepoda). Int J Parasitol. PMID: 19445947 Farlora, R., Araya, J. y Gallardo, C. 2014. Discovery of sex-related genes by high-throughput transcriptome sequencing from the salmon louse Caligus rogercresseyi. Marine Genomics. Doi: 10.1016/j.margen.2014.02.005. Filatov, D.A. 2002. Proseq: software for preparation and evolutionary analysis of DNA sequence datasets. Mol Ecol Notes 2: 621–624. Folmer, O., Black M., Hoeh W., Lutz R. y Vrijenhoek R. 1994. DNA primers for amplification of mitochondrial cytochrome coxidase subunit I from diverse metazoan invertebrates. Mol. Mar. Biol. Biotechnol. 3: 294-299. Gallardo, C., Valenzuela, V., Núñez, G. 2013a. RNA-seq analysis using de novo assembled transcriptome from salmon lice Caligus rogercresseyi. PlosOne. Doi: 10.1371/journal.pone. 0092239. Gallardo, C., Valenzuela, V., Núñez, G., Chávez, J., Maldonado, W. 2013b. Transcriptome analysis of couch potato (CPO) protein reveals expression pattern associated to early development in salmon lice Caligus rogercresseyi. Gene. Doi: 10.1016/j.gene.2013.11.100. Gharbi, K., Glover, K.A., Stone, L.C., MacDonald, E.S., Matthews, L., Grimholt, U., Stear, M.J. 2009. Genetic dissection of MHC-associated susceptibility to Lepeophtheirus salmonis in Atlantic salmon. BMC Genetics 10, 20. Glover, K.A., Grimholt, U., Bakke, H.G., Nilsen, F., Storset, A., Skaala, O. 2007. Major histocompatibility complex (MHC) variation and susceptibility to the sea louse Lepeophtheirus salmonis in Atlantic salmon Salmo salar. Diseases of Aquatic Organisms, 76, 57-65. Gonçalves, A. T., Farlora, R. y Gallardo C. 2013. Transcriptome survey of lipid metabolic pathways involved in energy production and ecdysteroids synthesis of the salmon louse Caligus rogercresseyi (Crustacea: Copepoda). Comparative Biochemistry and Physiology B. Doi: 10.1016/j.cbpb.2014.07.002. González MT, Molinet C, Arenas B, Asencio G, Carvajal J. 2012ñnFecundity of sea louse Caligus rogercresseyi on native host Eleginops maclovinus captured around salmon farms from Southern Chile. Aquacult Res, 43 (6):853-60 Himelreichs, J. 2013. Variabilidad del gen ribosomal 18S en Caligus rogercresseyi presente en Salmo salar y Eleginops maclovinus provenientes del ecosistema sur-austral de Chile. Seminario titulo de Ingeniería en Biotecnología Marina y Acuicultura, Univ. de Concepción, 67 pp. Isdal, E., Nylund, A., y Navdal, G. 1997. Genetic differences among salmon lice (Lepeophtheirus salmonis) from six Norwegian coastal sites: evidence from allozymes. Bulletin of the European Association of Fish Pathologists, 17: 17-22 Larkin, MA, Blackshields G, Brown NP, Chenna R, McGettigan PA, McWilliam H, Valentin F, Wallace, IM, Wilm A, Lopez R, Thompson JD, Gibson TJ, Higgins DG. 2007. Clustal W and Clustal X version 2.0. Bioinformatics, 23, 2947-2948. Leung, TLF, Donald KM, Keeney DB, Koehler AV, Peoples RC, Poulin R. 2009. Trematode parasites of Otago Harbour (New Zealand) soft-sediment intertidal ecosystems: life cycles, ecological roles and DNA barcodes. NZJ Mar Freshwater Res 43:857-65. Maldonado, W. y Gallardo C. 2013. Increased transcriptional response of SERPINs evidences a putative mechanism of immune evasion in the salmon louse Caligus rogercresseyi. Marine Genomics. Doi: 10.1016/j.margen.2014.04.006. McKay, S.J., Devlin R.H. & Smith M.J. 1996. Phylogeny of Pacific salmon and trout based on growth hormone type-2 and mitochondrial NADH dehydrogenase subunit 3 DNA sequences. Canadian Journal of Fisheries and Aquatic Sciences 53, 1165–1176. Morales, F.N., Hernández Z.L., Gómez S., Pérez G. 2013: Redescription of Caligus serratus Shiino, 1965 (Copepoda: Caligidae) parasitic on eleven fish species from Chamela Bay in the Mexican Pacific. Acta Parasitol 58(3): 367–375 Muñoz G. y Olmos V. 2007. Revisión bibliográfica de especies ectoparásitas y hospedadoras de sistemas acuáticos de Chile. Rev Biol Mar Oceanogr 42 (2):89-148. Nagasawa, K. 2004. Sea lice, Lepeophtheirus salmonis and Caligus orientalis (Copepoda: Caligidae), of wild and farmed fish in sea and brackish waters of Japan and adjacent regions: A review. Zool Stud 43:173-178 Nagasawa, K, Takami T. 1993. Host utilization by the salmon louse Lepeophtheirus salmonis (Copepoda, Caligidae) in the sea of Japan. J Parasitol 79:127-130 Núñez, G. y Gallardo C. 2013. High-throughput SNP discovery and transcriptome expression profiles from the salmon louse Caligus rogercresseyi (Copepoda: Caligidae). Comparative Biochemistry and Physiology D. Doi: 10.1016/j.cbd.2014.01.003. Núñez, G., Valenzuela, V., Pino Marambio, J., S. Wadsworth y C. Gallardo. 2014. Insights into the olfactory system in the ectoparasite Caligus rogercresseyi: Molecular characterization and gene transcription analysis of novel ionotropic receptors. Experimental Parasitology. In press. Oines, O. y Heuch P.A. 2005. Identification of sea louse species of the genus Caligus using mtDNA. J Mar Biol Ass UK 85: 73–79. Song, Y, Wang GT, Yao WJ, Gao Q, Nie P. 2008. Phylogeny of freshwater parasitic copepods in the Ergasilidae (Copepoda: Poecilostomatoida) based on 18S and 28S rDNA sequences. Parasitol Res 102:299-306. Swofford, D.L. 2003. PAUP*. Phylogenetic analysis using parsimony (*and other methods). [Version 4b]. Sunderland, Massachusetts: Sinauer Associates. Tamura, K, Peterson D, Peterson N, Stecher G, Nei M y Kumar S. 2011. MEGA5: Molecular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods. Molecular Biology and Evolution 28: 2731-2739. Tamura, K, Stecher G, Peterson D, Filipski A, Kumar S. 2013. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0. Molecular Biology and Evolution, 30, 2725-2729. Tjensvoll, K, Hodneland K, Nilsen F, Nylund A. 2005. Genetic characterization of the mitochondrial DNA from Lepeophtheirus salmonis (Crustacea : Copepoda). A new gene organization revealed. Gene 353:218-230 Todd, CD, Walker AM, Wolff K, Northcott SJ, Walker AF, Ritchie MG, Hoskins R, Abbott RJ, Hazon N. 1997. Genetic differentiation of populations of the copepod sea louse Lepeophtheirus salmonis (Kroyer) ectoparasitic on wild and farmed salmonids around the coasts of Scotland: Evidence from RAPD markers. J Exp Mar Biol Ecol 210:251-274 Todd, CM, Walker AM, Ritchie MG, Graves JA, Walker AF. 2004. Population genetic differentiation of sea lice (Lepeophtheirus salmonis) parasitic on Atlantic and Pacific salmonids: analyses of microsatellite DNA variation among wild and farmed hosts. Can J Fish Aquat Sci 61:1176-1190 Valenzuela, V. y Gallardo C. 2013a. Molecular characterization and transcription analysis of P-glycoprotein gene from the salmon louse Caligus rogercresseyi. Aquaculture Research and Development. Doi: 10.4172/2155-9546.1000236. Valenzuela, V. y Gallardo C. 2013b. TLR and IMD signaling pathways from Caligus rogercresseyi (Crustacea: Copepoda): In silico gene expression and SNPs discovery. Fish and Shellfish Immunology. Doi: 10.1016/j.fsi.2013.12.019. Yasawa, R M. Yasuike, J. Leon, G. Cooper, M Beetz-Sargent, A. Robb. R. Holt, R. Moore, W. Davidson, S. Jones, F. Koop. 2008. EST and mitochondrial DNA sequences support a distinct Pacific form of salmon louse, Lepeophtheirus salmonis. Marine Biotechnology 10: 741-749.
















