Desarrollan PCR-RFLP que permite diferenciar entre genogrupos de Piscirickettsia
Chile: Creado por investigadores chilenos, el nuevo PCR basado en el gen 16S permitiría una posterior diferenciación de los genogrupos LF-89 o EM-90 de P. salmonis mediante enzimas de restricción.
Históricamente, los aislados de Piscirickettsia salmonis se han clasificado dentro de los genogrupos LF-89 o EM-90, donde evidencia reciente apunta a que también podría existir un tercer genogrupo.
Al descubrirse que entre los genogrupos pueden existir diferencias importantes respecto de su patogenicidad y respuestas inmunes, la diferenciación rápida entre las especies ha cobrado valor.
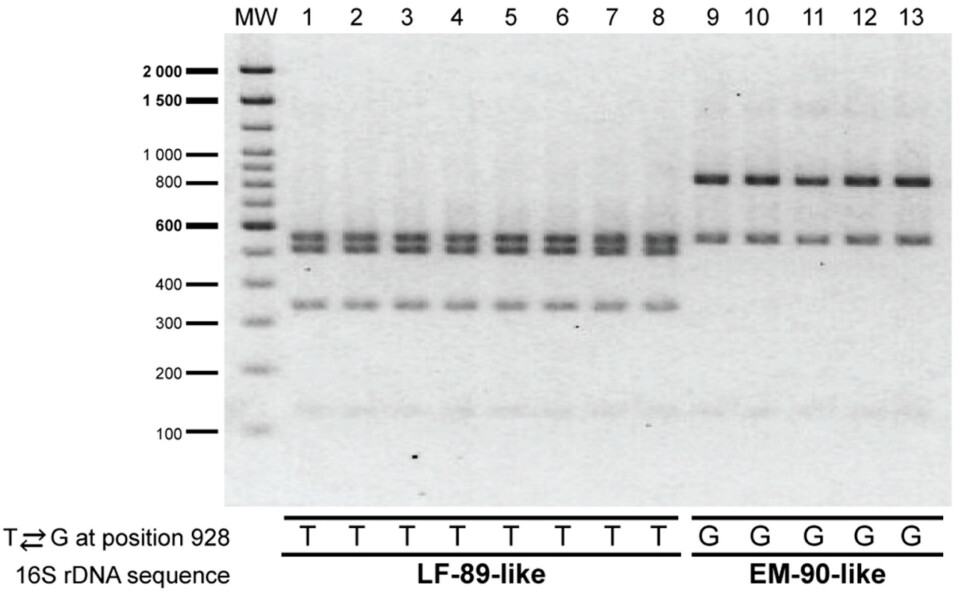
Por esta razón, científicos chilenos desarrollaron un PCR-RFLP (Polimorfismos de longitud de fragmentos de restricción) para la amplificación del gen ARNr 16S de ambos genogrupos del patógeno y otras bacterias, para luego lograr la diferenciación de las especies mediante enzimas de restricción.
Para el diseño de los partidores del PCR-RFLP, los expertos utilizaron 76 secuencias publicadas, 38 de cada especie. Luego, para la validanción experimental, compararon los patrones de restricción de 13 cepas de P. salmonis, para continuar con 57 muestras de campo desconocidas obtenidas de los tejidos de peces muertos o moribundos.
Con respecto a la enzima “XapI” seleccionada para lograr una correcta y fácil deferenciación de los genogrupos, los autores del estudio señalaron que se generan “tres bandas informativas y fácilmente reconocibles en el fragmento del gen 16S ARNr de las cepas EM-90-like (840, 548 y 119 pb) y cuatro en el fragmento del gen 16S ADNr de las cepas LF-89-like (548, 509, 331 y 119 pb)”.

Los expertos también señalaron que en las muestras de campo, a pesar de la presencia de mezclas de ADN de distintas bacterias, el patrón de digestión característico de los genogrupos de P. salmonis se pudo detectar sin necesidad de un cultivo microbiológico y aislamiento bacteriano.
“En este estudio, desarrollamos con éxito un método simple de genotipado para identificar los genogrupos de P. salmonis. Nuestro ensayo PCR-RFLP es un método rápido y rentable, que permite diferenciar claramente los genogrupos en las cepas de P. salmonis y también en las muestras de campo”, concluyeron los investigadores chilenos.
Lea el artículo completo titulado “PCR-RFLP Detection and Genogroup Identification of Piscirickettsia salmonis in Field Samples” aquí.




















